Uriel Garduño-Montes de Oca*
Universidad Nacional Autónoma de México, Instituto de Biología, Colección Nacional de Helmintos, Apartado postal 70-153, 04510 Ciudad de México, México
*Autor para correspondencia a: edurgam@ciencias.unam.mx (U. Garduño-Montes de Oca)
Recibido: 22 agosto 2022; aceptado: 16 marzo 2023
http://zoobank.org/urn:lsid:zoobank.org:pub:ACFD46B7-C983-4C8C-BCD2-B9B8983A8C40
Resumen
Se presenta la descripción de Lepidonema zuritai n. sp. y Phalacronema vegae n. sp. (Nematoda, Hystrignathidae), del intestino de Heliscus tropicus Percheron, 1835 y Passalus cognatus Truqui, 1857 (Passalidae), recolectados en Tamaulipas, noreste de México. Las especies nuevas son descritas con base en las características morfológicas de la anatomía interna y externa típicamente utilizadas para la caracterización taxonómica de histrignátidos, como el número de ovarios, forma del esófago y sus partes, la presencia, forma y disposición de ornamentaciones cuticulares (espinas, alas, estriaciones), entre otras. Adicionalmente, se presentan las relaciones filogenéticas de las especies descritas, a partir de caracteres moleculares obtenidos de secuencias parciales de las subunidades menor y mayor del ribosoma, marcadores informativos típicamente usados en filogenética de nemátodos. Ambas especies se recuperaron dentro del clado que agrupa a la mayoría de los géneros de Hystrignathidae analizados y, aunque no son del todo claras las relaciones al interior del mismo, los resultados obtenidos sugieren un parentesco estrecho entre Lepidonema zuritai n. sp. y Xyo pseudohystrix, mientras que las relaciones entre Phalacronema vegae n. sp. al interior del clado aún no son claras.
Palabras clave: Nemátodos; Oxiuros; Arthropoda; Escarabajos de madera; Sistemática molecular; Taxonomía
© 2023 Universidad Nacional Autónoma de México, Instituto de Biología. Este es un artículo Open Access bajo la licencia CC BY-NC-ND
(http://creativecommons.org/licenses/by-nc-nd/4.0/).
Two new species of Hystrignathidae (Nematoda: Thelastomatoidea) associated to Heliscus tropicus and Rhodocanthopus cognatus (Coleoptera: Passalidae) from Tamaulipas, Mexico
Abstract
The description of Lepidonema zuritai n. sp. and Phalacronema vegae n. sp. (Nematoda, Hystrignathidae, from the intestine of Heliscus tropicus Percheron, 1835 and Passalus cognatus Truqui, 1857 (Passalidae), collected in Tamaulipas, northeastern Mexico, is presented. The new species are described based on the morphological characteristics of the internal and external anatomy typically used for the taxonomic characterization of histrignatids, such as number of ovaries, shape of the esophagus and its parts, presence, shape and disposition of cuticular ornamentations (spines, wings, striations), among others. Additionally, phylogenetic relationships of the described species are presented, based on molecular characters obtained from partial sequences of the minor and major subunits of the ribosome, informative markers typically used in nematode phylogenetics. Both species were recovered within the clade that groups most of Hystrignathidae genera analyzed, and although relationships within the clade are not entirely clear, results obtained suggest a close relationship between Lepidonema zuritai n. sp. and Xyo pseudohystrix, while relationships of Phalacronema vegae n. sp. within the clade are still unclear.
Keywords: Nematodes; Pinworms; Arthropoda; Wood beetles; Molecular systematics; Taxonomy
© 2023 Universidad Nacional Autónoma de México, Instituto de Biología. This is an open access article under the CC BY-NC-ND license
Introducción
Los nemátodos han colonizado diversos ambientes y establecido interacciones biológicas con una amplia variedad de organismos. Específicamente, el parasitismo ha sido una estrategia exitosa que ha surgido en diferentes linajes a lo largo de la historia del phylum Nematoda (Blaxter et al., 1998). Los grupos de nemátodos Oxyuridomorpha, Drilonematomorpha y Ascaridomorpha son considerados ancestrales dentro de los nemátodos parásitos de invertebrados terrestres. Particularmente, Oxyuridomorpha agrupa a varias especies de ciclo de vida directo ampliamente estudiados desde una perspectiva médica y veterinaria, ya que algunas especies, como Enterobius vermicularis Linnaeus, 1758 y Oxyuris equi Schrank, 1788 son relevantes para la salud humana y equina, respectivamente. Estas 2 especies de nemátodos, así como otros oxiuros asociados a vertebrados conforman un grupo de alrededor de 700 especies clasificadas dentro de la superfamilia Oxyuroidea (Hodda, 2007). No obstante, otras 2 superfamilias también pertenecen a Oxyuridomorpha: Thelastomatoidea con alrededor de 450 especies y Coronostomatoidea con alrededor de 50 especies, ambas agrupan nemátodos asociados a Arthropoda, lo que permite suponer un espectro más amplio de hospederos asociados a estas 2 superfamilias, posiblemente más diversas que aquella relacionada exclusivamente con vertebrados (Adamson y Van Waerebeke, 1992; Carreno, 2014; Garduño-Montes de Oca et al., 2020; Hodda, 2007).
A pesar de lo anteriormente planteado, parece haber un sesgo en la descripción de oxiuros, que puede ser explicado a partir del interés médico y veterinario que ha llevado a estudiar con mayor urgencia a aquellos nemátodos asociados con vertebrados, lo que contrasta con los pocos estudios enfocados en oxiuros asociados a artrópodos, la mayoría de ellos dirigidos solo en algunos grupos de hospederos, entre los que destacan dípteros, ortópteros, coleópteros, blatodeos y miriápodos (Carreno, 2014). Afortunadamente, en los últimos años ha habido un interés creciente en estudiar a este grupo particular de oxiuros, lo que ha resultado en el descubrimiento de nuevos taxones, particularmente en América y Asia (Morffe et al., 2021; Zhang et al., 2021).
El presente estudio aporta conocimiento sobre la superfamilia Thelastomatoidea, mediante la descripción de 2 especies nuevas de Hystrignathidae encontradas en los escarabajos de madera Heliscus tropicus y Rhodocanthopus cognatus en Tamaulipas. Esta familia de nemátodos agrupa especies asociadas a coleópteros de la familia Passalidae que concentra alrededor de 90 especies, muchas de ellas endémicas, y forma un grupo de escarabajos particularmente diverso en México, pero poco estudiado desde un enfoque parasitológico (Reyes-Castillo, 2000). Se presentan las características morfológicas de los nuevos taxones, así como un análisis de sus relaciones filogenéticas a nivel de familia, con base en caracteres moleculares.
Materiales y métodos
En total, se recolectaron 27 individuos de Passalus cognatus y 26 de Heliscus tropicus en sitios con una vegetación característica del bosque mesófilo de montaña, dentro de la Reserva de la Biosfera El Cielo y a una elevación de 200-1,800 m snm. Los hospederos se recolectaron de forma directa sobre troncos caídos o con la ayuda de herramientas de jardinería, fueron preservados en etanol al 96% para su examinación posterior en el laboratorio. Los pasálidos se identificaron con la ayuda de claves taxonómicas y literatura especializada para el grupo (Reyes-Castillo, 1970), una vez identificados por el personal de la Colección Entomológica IEXA del Instituto de Ecología, A.C. (INECOL), fueron entregados a dicha institución. La disección de los escarabajos recolectados se llevó a cabo bajo un microscopio estereoscópico, se extrajo el intestino a través del retiro del abdomen; posteriormente, se revisaron internamente las diferentes secciones del tubo digestivo. Los nemátodos recuperados se preservaron en etanol al 96% para su posterior estudio morfológico y molecular.
Los nemátodos encontrados fueron examinados para su identificación taxonómica a partir de su caracterización morfológica. Para esto, algunos ejemplares se transparentaron con una solución de glicerina diluida en etanol al 96% en una proporción de 1:1. Una vez transparentados, los nemátodos fueron observados en el microscopio óptico compuesto; así mismo, se obtuvieron características morfométricas típicamente utilizadas en el estudio taxonómico de nemátodos (tabla 1) y se realizaron ilustraciones con ayuda de una cámara clara montada en un microscopio óptico Olympus BX53; las medidas son presentadas en milímetros (a menos que se indique otra unidad) en el siguiente orden: intervalo (promedio ± desviación estándar, n = número de individuos medidos). Las proporciones propuestas por De Man (1884) para la caracterización morfométrica de nemátodos fueron calculadas de la siguiente forma: a = longitud total del cuerpo/ ancho máximo del cuerpo, b = longitud total del cuerpo/ longitud total del esófago, c = longitud total del cuerpo/ longitud de la cauda, c’= longitud de la cauda/ancho del cuerpo a nivel del ano, V% = posición de la vulva expresada como porcentaje de la longitud total del cuerpo y V’% = posición de la vulva expresada como porcentaje de la distancia desde el extremo anterior hasta el ano. El material tipo fue depositado en la Colección Nacional de Helmintos (CNHE) del Instituto de Biología, Universidad Nacional Autónoma de México (IB-UNAM).
Un ejemplar de cada especie de nemátodo fue procesado en el Laboratorio de Microscopía y Fotografía de la Biodiversidad del Laboratorio Nacional de Biodiversidad (LANABIO) IB-UNAM, para lo cual primero fueron limpiados con detergente biológico y vibración mecánica. Se deshidrataron a punto crítico con dióxido de carbono mediante su inmersión en etanol a 96% y 100% durante 2 h en cada concentración, utilizando un desecador Emitech, K850. Posteriormente, los ejemplares fueron montados y se cubrieron con una mezcla de oro y paladio en el sistema de revestimiento Quorum Q150R. Finalmente, las muestras fueron examinadas a 10 kV en un microscopio Hitachi Stereoscan modelo SU1015.
Para el análisis de inferencia filogenética, se utilizaron caracteres moleculares obtenidos a partir de secuencias de nucleótidos de ADN. La obtención de secuencias se realizó mediante reacciones de amplificación y secuenciación de 2 fragmentos de genes ribosomales: la subunidad mayor (28S o LSU) y menor (18S o SSU) del ribosoma, marcadores típicamente usados en el estudio de las relaciones filogenéticas de nemátodos. Tres ejemplares de Lepidonema zuritai n. sp. y 2 de Phalacronema vegae n. sp. fueron fijados en etanol 96% y refrigerados a -20°C hasta la extracción de ADN genómico, la cual se realizó con los reactivos de extracción de DNeasy ® Blood and Tissue (Qiagen, USA). Los fragmentos de los genes mencionados fueron obtenidos a partir de su amplificación mediante reacciones en cadena de la ADN polimerasa (PCR por sus siglas en inglés). Cada reacción estuvo compuesta por 0.2 µl de cada uno de los 2 oligonucleótidos, a una concentración de 10 µM, 0.1 µl de la ADN polimerasa MyTaq polymerase (Bioline, UK) y a una de 0.5 U/µl, 3 MyTaq polymerase (Bioline, UK) de la solución amortiguadora 5x PCR buffer proporcionada con la enzima, y 3 µl de ADN genómico en un volumen total de 15 µl por reacción. El fragmento de la región D2-D3 del gen de la subunidad mayor se amplificó con el par de oligonucleótidos D2A (5´–ACAAGTACCGTGAGGGAAAGTTG–3´) y D3B (5´–TCGGAAGGAACCAGCTACTA–3´) (Nunn, 1992). Por su parte, los fragmentos del gen de la unidad menor ribosomal fueron amplificados con el par de oligonucleótidos nSSU_F_04 (5´–GCTTGTCTCAAAGATTAAGCC–3´) (Blaxter et al., 1998) y nSSU_R_82 (5´–TGATCCTTCTGCAGGTTCACCTAC–3´) (Medlin et al., 1988). El perfil térmico para las reacciones incluyó una desnaturalización inicial a 94°C durante 5 min seguida durante 35 ciclos de 94 °C (98 para LSU) durante 30 s, 54 °C (50 para LSU) durante 30 s y 72 °C durante 60 s, seguido por una fase de extensión post amplificación a 72 °C durante 7 min. Los productos de las reacciones de PCR fueron secuenciados en ambas direcciones, utilizando los mismos oligonucleótidos que se usaron en la reacción de amplificación para la región D2-D3, y se utilizaron los oligonucleótidos 988F (5´–CTCAAAGATTAAGCCATGC–3´) y 1912R (5´–TTTACGGTCAGAACTAGGG–3´) para la secuenciación de la región 5´ del gen SSU (Holterman et al., 2006). Estas técnicas de biología molecular fueron implementadas en el Laboratorio de Secuenciación Genómica de la Biodiversidad y de la Salud, Instituto de Biología, Universidad Nacional Autónoma de México (IBUNAM). La secuencia consenso de cada individuo procesado fue ensamblada a partir de la lectura de los histogramas con ayuda del software Geneious Prime® 2019.2.3 (http://www.geneious.com, Kearse et al., 2012). Todas las secuencias obtenidas en este estudio fueron depositadas en GenBank (National Center for Biotechnology Information; NCBI).
Tabla 1
Características morfométricas de Lepidonema zuritai n. sp. y Phalacronema vegae n. sp. expresadas en milímetros
| Carácter | Lepidonema zuritai n. sp. | Phalacronema vegae n. sp. | ||||||
| Holotipo | Paratipos | Holotipo | Paratipos | |||||
| Rango | Promedio | n | Rango | Promedio | n | |||
| a = Longitud total del cuerpo / ancho máximo del cuerpo | 21.540 | 21.518-23.414 | 22.045 ± 0.716 | 6 | 6.676 | 6.184-6.676 | 6.449 ± 0.248 | 3 |
| b = Longitud total del cuerpo / longitud total del esófago | 6.290 | 5.488-6.290 | 5.927 ± 0.347 | 5 | 5.787 | 5.599-6.133 | 5.840 ± 0.271 | 3 |
| c = Longitud total del cuerpo / longitud de la cauda | 8.998 | 7.394-9.690 | 8.603 ± 0.860 | 6 | 60.930 | 50.522-60.930 | 54.440 ± 5.661 | 3 |
| c’ = Longitud de la cauda / ancho del cuerpo a nivel del ano | 5.267 | 4.625-5.414 | 5.079 ± 0.268 | 6 | 0.500 | 0.447-0.543 | 0.497 ± 0.048 | 3 |
| V% = Posición de la vulva expresada como porcentaje de la longitud total del cuerpo | 54.572 | 50.479-58.160 | 54.795 ± 2.891 | 6 | 63.563 | 61.702-63.563 | 62.572 ± 0.936 | 3 |
| V’% = Posición de la vulva expresada como porcentaje de la distancia desde el extremo anterior hasta el ano | 61.396 | 57.337-65.221 | 62.058 ± 2.834 | 6 | 64.623 | 62.915-64.623 | 63.750 ± 0.855 | 3 |
| Longitud total | 3.085 | 2.439-3.112 | 2.856 ± 0.286 | 8 | 2.248 | 2.139-2.302 | 2.230 ± 0.083 | 3 |
| Localización de la vulva con respecto al extremo anterior | 1.684 | 1.283-1.784 | 1.568 ± 0.210 | 8 | 1.429 | 1.320-1.438 | 1.395 ± 0.066 | 3 |
| Inicio de alas laterales con respecto al extremo anterior | – | – | – | – | 0.273 | 0.246-0.273 | 0.264 ± 0.016 | 3 |
| Final de alas laterales con respecto al extremo posterior | – | – | – | – | 0.119 | 0.109-0.119 | 0.116 ± 0.006 | 3 |
| Ancho anillo cuticular anterior | 0.033 | 0.028-0.037 | 0.031 ± 0.003 | 8 | 0.033 | 0.033-0.033 | 0.033 ± 0.000 | 3 |
| Largo anillo cuticular anterior | 0.009 | 0.009-0.013 | 0.011 ± 0.002 | 8 | 0.011 | 0.009-0.011 | 0.010 ± 0.001 | 3 |
| Final de espinas cefálicas con respecto al poro excretor | 0.130 | 0.087-0.141 | 0.113 ± 0.024 | 7 | – | – | – | – |
| Localización del ano con respecto al extremo posterior | 0.343 | 0.321-0.343 | 0.332 ± 0.008 | 8 | 0.037 | 0.037-0.046 | 0.041 ± 0.004 | 3 |
| Localización del ano con respecto al extremo anterior | 2.742 | 2.109-2.791 | 2.524 ± 0.285 | 8 | 2.211 | 2.097-2.257 | 2.188 ± 0.082 | 3 |
| Longitud total del estoma | 0.054 | 0.050-0.054 | 0.053 ± 0.002 | 8 | 0.028 | 0.028-0.030 | 0.029 ± 0.001 | 3 |
| Ancho del cuerpo a nivel de la vulva | 0.143 | 0.104-0.143 | 0.130 ± 0.015 | 7 | 0.337 | 0.337-0.355 | 0.346 ± 0.009 | 3 |
| Ancho del cuerpo a nivel del ano | 0.065 | 0.063-0.069 | 0.065 ± 0.002 | 8 | 0.074 | 0.074-0.102 | 0.084 ± 0.016 | 3 |
| Localización del anillo nervioso con respecto al extremo anterior | 0.263 | 0.239-0.276 | 0.261 ± 0.016 | 8 | 0.191 | 0.191-0.206 | 0.197 ± 0.008 | 3 |
| Ancho del cuerpo a nivel del anillo nervioso | 0.104 | 0.093-0.104 | 0.101 ± 0.004 | 8 | 0.217 | 0.195-0.217 | 0.204 ± 0.011 | 3 |
| Distancia del extremo anterior al poro excretor | 0.774 | 0.728-0.828 | 0.772 ± 0.038 | 8 | 0.308 | – | – | 1 |
| Ancho del cuerpo a nivel de poro excretor | 0.132 | 0.117-0.132 | 0.125 ± 0.007 | 8 | 0.246 | – | – | 1 |
| Longitud del procorpus | 0.380 | 0.369-0.397 | 0.386 ± 0.012 | 8 | 0.280 | 0.273-0.280 | 0.278 ± 0.004 | 3 |
| Longitud de itsmo | 0.046 | 0.043-0.050 | 0.046 ± 0.003 | 8 | 0.022 | 0.015-0.022 | 0.017 ± 0.004 | 3 |
| Longitud del bulbo esofágico | 0.065 | 0.059-0.065 | 0.064 ± 0.003 | 8 | 0.087 | 0.087-0.087 | 0.087 ± 0.000 | 3 |
| Longitud del esófago | 0.490 | 0.471-0.506 | 0.496 ± 0.015 | 8 | 0.388 | 0.375-0.388 | 0.382 ± 0.007 | 3 |
| Ancho del procorpus en la base | 0.039 | 0.037-0.041 | 0.039 ± 0.002 | 8 | 0.043 | 0.043-0.043 | 0.043 ± 0.000 | 3 |
| Ancho del procorpus su extremo anterior | 0.026 | 0.022-0.028 | 0.026 ± 0.003 | 8 | 0.028 | 0.028-0.030 | 0.029 ± 0.001 | 3 |
| Ancho de itsmo | 0.026 | 0.022-0.026 | 0.025 ± 0.002 | 8 | 0.026 | 0.026-0.033 | 0.029 ± 0.003 | 3 |
| Ancho del bulbo | 0.065 | 0.061-0.067 | 0.064 ± 0.003 | 8 | 0.091 | 0.091-0.093 | 0.092 ± 0.001 | 3 |
| Longitud de huevos | 0.106 | 0.102-0.106 | 0.104 ± 0.002 | 5 | 0.137 | 0.130-0.139 | 0.135 ± 0.005 | 3 |
| Ancho de huevos | 0.037 | 0.035-0.039 | 0.037 ± 0.002 | 8 | 0.067 | 0.067-0.069 | 0.068 ± 0.001 | 3 |
Con la finalidad de explorar las relaciones filogenéticas de los nuevos taxones aquí descritos, se analizaron diversas secuencias disponibles en GenBank. Se incluyeron secuencias de especies asignadas a Thelastomatoidea y Coronostomatoidea, así como de algunas especies de Oxyuroidea. El análisis filogenético se realizó con los 2 marcadores moleculares especificados en la sección anterior, los alineamientos se realizaron de manera independiente y posteriormente fueron concatenados. El conjunto de secuencias generadas y recopiladas de cada gen se alinearon utilizando el método L-INS-I en el programa MAFFT versión 7 (Katoh et al., 2019) con un ajuste manual en el programa Mesquite v. 3.6 (Maddison y Maddison, 2019). Los espacios en las secuencias generados por la alineación fueron tratados como datos faltantes. El programa jModeltest v. 3.0 (Posada y Crandall, 1998) se utilizó para inferir el modelo de evolución con mejor ajuste según el criterio de información de Akaike. La inferencia filogenética se realizó utilizando los métodos de optimización de Inferencia Bayesiana (IB) y Parsimonia. El análisis de IB se realizó en el programa MrBayes v. 3.2.1 (Ronquist et al., 2012), los ajustes para este análisis se establecieron de la siguiente forma: 2 análisis simultáneos con 4 cadenas de Markov Monte Carlo (MCMC) durante 107 generaciones, un muestreo cada 100 generaciones, descartando 25% como “burn-in”. Los valores estadísticos de convergencia fueron revisados utilizando el programa Tracer v. 1.7.1 (Rambaut et al., 2018). Finalizado el análisis, a partir del muestreo de árboles, se calcularon los valores de probabilidad posterior de cada nodo y se construyó un árbol de consenso con una regla de mayoría de 50%. Por otro lado, el análisis de parsimonia se realizó en el programa TNT v. 1.1 (Goloboff et al., 2008), mediante una búsqueda heurística tradicional con los espacios en las secuencias alineadas considerados como datos faltantes, el soporte de nodos se calculó mediante un remuestreo de bootstrap utilizando 10,000 pseudoréplicas. Los resultados de ambos análisis se ilustraron en un árbol filogenético construido en el programa FigTree v. 1.4.2 (Rambaut, 2006).
Descripción
Lepidonema zuritai n. sp. (fig. 1)
http://zoobank.org/urn:lsid:zoobank.org:act:77159114-0F5F-4973-99F3-E9BEC3DB13C9
Diagnosis (basada en 6 individuos). Hembras grávidas. Cuerpo fusiforme y esbelto (fig. 1A). Sin alas laterales. La región anterior presenta 2 anillos cuticulares, en la región apical del anillo anterior se localizan 4 pares de papilas dispuestas simétricamente, 2 en la región ventral y 2 en la dorsal, así como 2 anfidios localizados lateralmente, el anillo posterior es poco evidente, no se encuentra dilatado ni presenta una longitud o anchura distinta a las filas transversales de espinas cuticulares que inician justo después de este anillo y terminan a una distancia considerablemente posterior al poro excretor (fig. 1A-C). Las filas transversales de espinas cuticulares se encuentran alineadas longitudinalmente, con las espinas dirigidas hacia la región posterior, las espinas de las primeras filas tienen forma de escama mientras que las subsecuentes tienen forma triangular, la primera fila presenta 20 espinas, las espinas van aumentando su tamaño hasta alcanzar un máximo a la mitad de su distribución, después de lo cual va disminuyendo (fig. 1B, C). El esófago es muscular, presenta un estoma ligeramente esclerosado en su luz, un procorpus dividido en el que la parte anterior es subcilíndrica, con el extremo anterior ligeramente menos ancho, la parte posterior del procorpus es notablemente más ancha que el resto del mismo, también es subcilíndrica y se une con el istmo cilíndrico, el cual se conecta a un bulbo esofágico subesférico, que presenta valvas esclerosadas en su interior (fig. 1A). El intestino es un poco más ancho que el bulbo en su extremo anterior, disminuyendo gradualmente su diámetro a lo largo del cuerpo (fig. 1A). El anillo nervioso se localiza ligeramente anterior a la división del procorpus, en la región media del mismo. Poro excretor localizado posterior al bulbo esofágico, distinguible por el cuerpo vesicular que rodea el conducto excretor (fig. 1A). El sistema reproductor es didelfo, anfidelfo, la vulva se localiza aproximadamente en la región media del cuerpo y en ambas ramas uterinas se observan huevos elipsoidales ornamentados con protuberancias pequeñas dispuestas en filas longitudinales en la superficie (fig. 1A, E). La cola es comparativamente corta, cónica, subulada (fig. 1A, D).
Resumen taxonómico
Hospedero tipo: Heliscus tropicus.
Sitio de infección: intestino medio.
Individuos depositados: Colección Nacional de Helmintos (CNHE), Instituto de Biología, UNAM; holotipo: CNHE 9759; paratipos: CNHE 9760 (5 individuos).
Localidad tipo: Reserva de la Biosfera El Cielo, Tamaulipas, México.
Etimología: el nombre específico está dedicado a la memoria de Martín Leonel Zurita García, amigo, colega e investigador dedicado al estudio de escarabajos en México.
Comentarios taxonómicos
El género Lepidonema fue propuesto por Cobb (1898) a partir de nemátodos encontrados en una larva de insecto no identificada en New South Wales, Australia; esta infección ha sido considerada accidental, ya que la mayoría de estos nemátodos infecta escarabajos pasálidos en estado adulto. Lepidonema bifurcata Cobb, 1898 corresponde a la especie tipo del género, al cual pertenecen otras 4 especies hasta la fecha: Lepidonema brasiliensis Travassos et Kloss, 1957 en Passalus punctiger punctatissimus de Brasil; Lepidonema caracae Kloss, 1962 en Passalus plicatus de Brasil; Lepidonema magnum Morffe et García, 2010 en Passalus interstitialis de Cuba; y Lepidonema teresae García, Ventosa et Morffe, 2009 en Passalus pertyi de Cuba (Adamson y Van Waerebeke, 1992; García et al., 2009; Morffe y García, 2010).
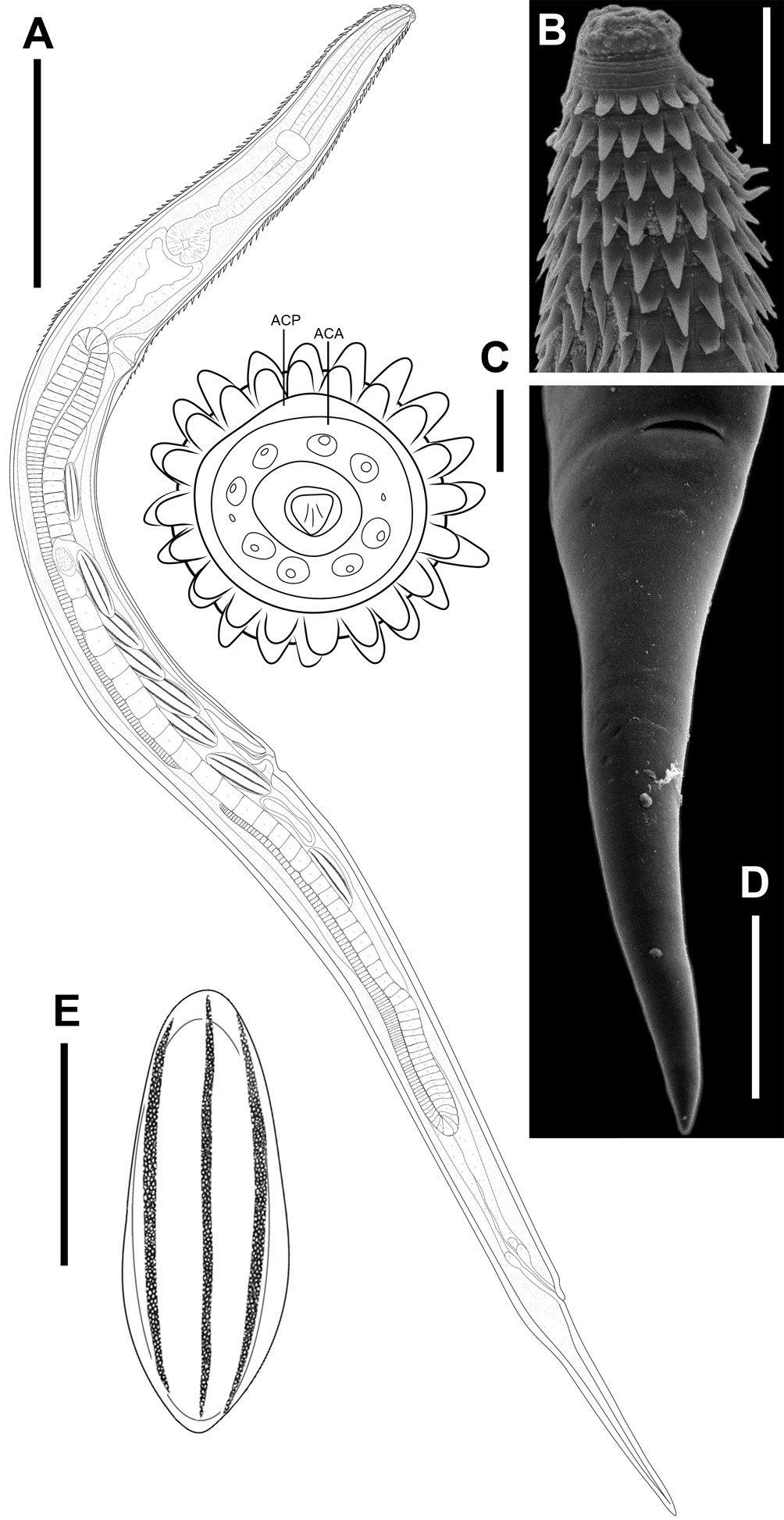
Las especies de Lepidonema se diferencian entre sí, principalmente, por la extensión de las espinas cuticulares, la distribución de las alas laterales, así como la morfología del apéndice caudal. De este modo, Lepidonema zuritai n. sp. puede distinguirse del resto de las especies, sobre todo, por la ausencia de ala lateral, la cual es exhibida con distinta extensión en el resto de las especies del género. Además, puede distinguirse de L. bifurcata, porque no presenta un apéndice caudal bifurcado, sino que es simple como en las otras especies de Lepidonema. Además de ésto, las espinas cuticulares en la especie nueva finalizan posterior al poro excretor, lo que contrasta con la distribución de las espinas en el resto de las especies, en las que suelen observarse con una distribución posterior máxima a la altura del bulbo esofágico. Lepidonema zuritai n. sp. es la primera especie del género descrita en México, así como la primera del género en la región neártica.
Phalacronema vegae n. sp. (fig. 2)
http://zoobank.org/urn:lsid:zoobank.org:act:79C96E20-55B8-41F5-AB9C-50B358AE0D52
Diagnosis (basada en 3 individuos). Hembras grávidas. Cuerpo robusto, ligeramente fusiforme (fig. 2A). Extremo anterior cónico, abruptamente truncado (fig. 2A, B). La región anterior del cuerpo presenta un anillo cuticular, donde se localizan 4 pares de papilas dispuestas simétricamente, 2 en la región ventral y 2 en la dorsal, así como 2 anfidios localizados lateralmente (fig. 2C). Sin espinas cuticulares (fig. 2A, B), pero con estriaciones cuticulares a lo largo del cuerpo (fig. 2B-D). El anillo nervioso se localiza aproximadamente a la mitad del esófago, cercano a la división del procorpus situada en la región media del mismo, el poro excretor se ubica a la altura del istmo, con un cuerpo vesicular comparativamente pequeño pero distinguible, la vulva se encuentra al inicio del tercio posterior del cuerpo (fig. 2A). El extremo posterior del cuerpo es obtuso, con un apéndice pequeño y romo, la apertura anal está cercana al extremo posterior (fig. 2A, D). Presentan alas laterales que inician a la mitad del procorpus y terminan un poco antes de la abertura anal (fig. 2A, B, D). El esófago es muscular, presenta un estoma comparativamente corto y ligeramente esclerosado en la luz del mismo, un procorpus dividido en una parte anterior subcilíndrica, con el extremo anterior ligeramente menos ancho, la parte posterior del procorpus es notablemente más ancha que el resto del procorpus, también es subcilíndrica, y se une con el istmo que es corto y de forma cilíndrica (fig. 2A). El bulbo esofágico es subesférico y presenta valvas esclerosadas en su interior (fig. 2A). El intestino es notablemente más ancho que el bulbo en su extremo anterior, disminuyendo gradualmente su diámetro a lo largo del cuerpo (fig. 2A). El sistema reproductor es didelfo, anfidelfo, con huevos elipsoidales ornamentados con pequeñas protuberancias dispuestas sin un orden aparente en la superficie (fig. 2E).
Resumen taxonómico
Hospedero tipo: Passalus cognatus.
Sitio de infección: intestino anterior.
Individuos depositados: Colección Nacional de Helmintos (CNHE), Instituto de Biología, UNAM; holotipo: CNHE 9767; paratipos: CNHE 9768 (2 individuos).
Localidad tipo: Reserva de la Biosfera El Cielo, Tamaulipas, México.
Etimología: el nombre específico está dedicado con singular afecto y gratitud a Viridiana Vega Badillo, investigadora dedicada al estudio de escarabajos.
Comentarios taxonómicos
Dentro de la familia Hystrignathidae, existen algunos géneros caracterizados por no presentar espinas cuticulares en la región anterior del cuerpo, como Anomalostoma Cordeiro, 1981, Anuronema Clark, 1978, Longior Travassos y Kloss, 1958, Klossnema Cordeiro, 1981, Ventelia Travassos y Kloss, 1958 y Phalacronema Clark, 1978, entre otros (Adamson y Van Waerebeke, 1992). Phalacronema es un género propuesto por Clark (1978) a partir de nemátodos encontrados en el intestino de escarabajos pasálidos de la especie Aulacocyclus mastersi, recolectados en el Parque Nacional Ku-ring-gai Chase, New South Wales, Australia. Hasta la fecha, solo se ha asignado una especie a este género, Phalacronema ardleyi Clark, 1978. Además de la falta de espinas cuticulares en la región anterior, Phalacronema se distingue de otros histrignátidos en que presenta un cuerpo comparativamente robusto y alargado, con un solo anillo cuticular en el extremo anterior del cuerpo (anillo cefálico), alas laterales bien desarrolladas y un esófago relativamente corto con un bulbo bien desarrollado e istmo subcilíndrico, claramente distinguibles (Adamson y Van Waerebeke, 1992; Clark, 1978). Phalacronema vegae n. sp. presenta algunas características que permiten distinguirla con claridad de P. ardleyi, principalmente la forma de la cola (extremo romo con un apéndice caudal pequeño vs. cónica sin apéndice caudal), el ala lateral comienza a la altura del corpus en ambas especies, pero termina posterior a la apertura anal en P. ardleyi y antes del ano en P. vegae n. sp. Además, el poro excretor se encuentra ubicado posterior al bulbo esofágico en P. ardleyi, mientras que en la nueva especie se encuentra antes de dicha estructura, específicamente a la altura del istmo. En P. vegae n. sp. los huevos presentan pequeñas protuberancias fungiformes, similares a las observadas en el género Triumphalisnema, pero no mencionadas por Clark (1978) en la descripción de P. ardleyi.
Phalacronema vegae n. sp. es la primera especie del género descrita en el continente americano; no obstante, una revisión detallada de la anatomía de P. ardleyi y particularmente de la ultraestructura de la superficie de los huevos, podría ser relevante para una caracterización más precisa del género.
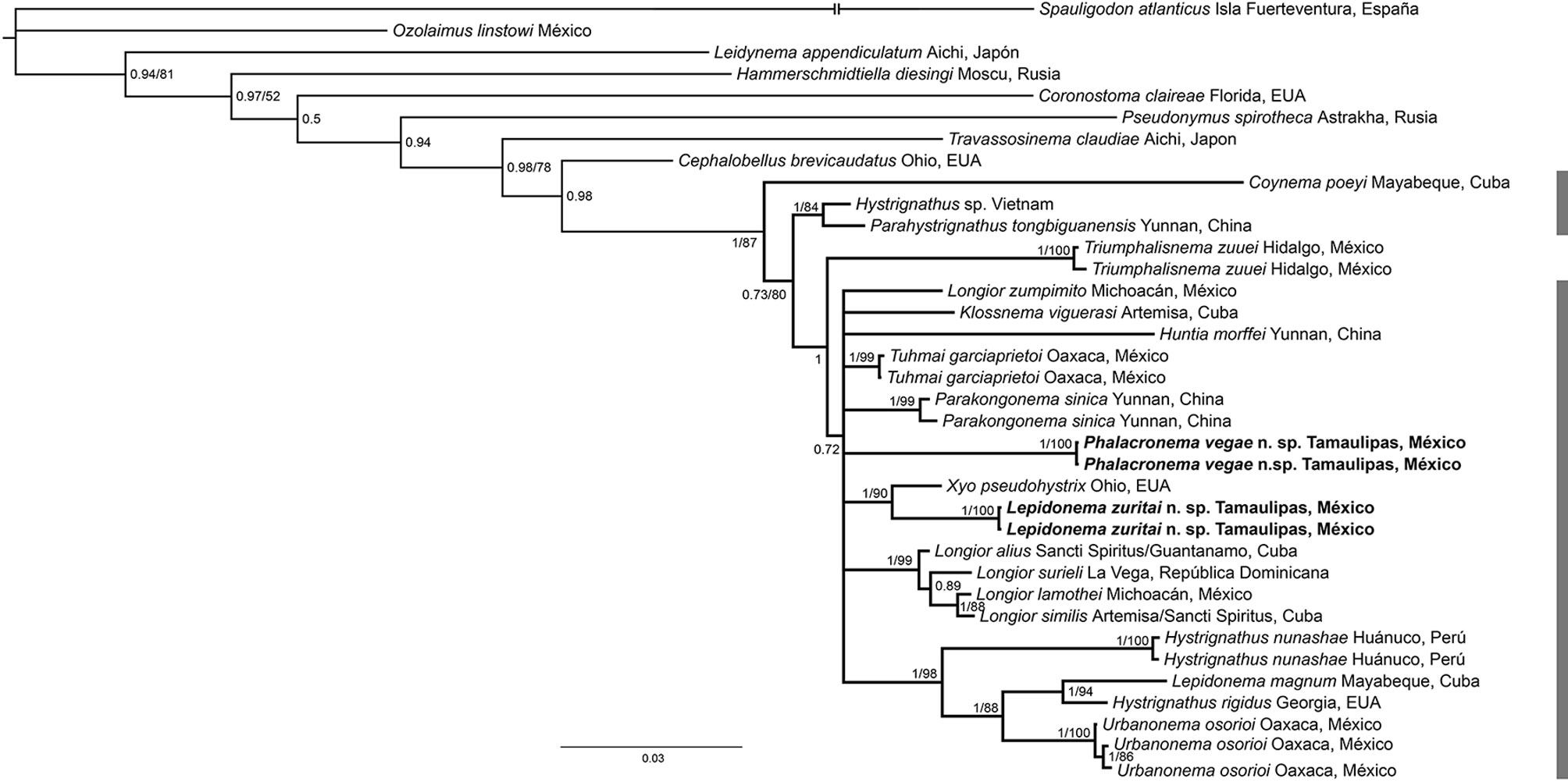
Resultados filogenéticos. Se obtuvieron secuencias nucleotídicas parciales de los genes SSU y LSU a partir de 2 individuos de cada una de las 2 especies descritas en el presente estudio, ambos marcadores moleculares fueron obtenidos para los individuos procesados. Las secuencias obtenidas fueron utilizadas para construir una matriz de secuencias concatenadas conformando un total de 36 terminales de diferentes especies asociadas en algún grado a Hystrignathidae (tabla 2), la matriz resultante del alineamiento tuvo una longitud de 3,007 sitios. Para el análisis de inferencia bayesiana se implementó el modelo GTR+G+I, de acuerdo con los resultados del análisis de jModeltest; los valores de convergencia fueron verificados en el programa Tracer V1.7.1. El análisis de parsimonia de la matriz concatenada resultó en un árbol de 1,900 pasos.
En general, las relaciones filogenéticas obtenidas por el análisis de parsimonia y de inferencia bayesiana resultaron congruentes entre sí, por lo que la hipótesis presentada en el árbol consenso es, en general, consistente (fig. 3). La familia Hystrignathidae se recuperó como grupo parafilético, con Triumphalisnema zuuei Garduño-Montes de Oca, Vega-Badillo, López-Caballero, Ramírez-Cañas y Mata-López, 2020 (Coronostomatoidea, Traklosiidae), anidada dentro del clado formado por todos los histrignátidos incluidos en el análisis (fig. 3). Consecuentemente, se recuperó a Coronostomatoidea como grupo parafilético, con Coronostoma claireae Phillips y Bernard, 2016 y Triumphalisnema zuuei posicionados en linajes separados. Adicionalmente, ambos métodos recuperaron la misma posición filogenética en el caso de Hystrignathus sp., Parahystrignathus tongbiguanensis Zhang, Yin, Carreno y Zhang, 2021 y Coynema poeyi Coy, García y Álvarez, 1993; así mismo, ambos métodos obtuvieron a las especies del género Longior formando un grupo parafilético y al clado conformado por Lepidonema zuritai n. sp. y Xyo pseudohystrix Travassos y Kloss, 1958. Por su parte, las relaciones de las especies Tuhmai garciaprietoi Garduño-Montes de Oca y Oceguera-Figueroa, 2020, Klossnema viguerasi Morffe, García, Hasegawa y Breugelmans, 2021, Phalacronema vegae n. sp., Longior zumpimito Morffe, García, Adams y Hasegawa, 2020, Huntia morffei Zhang, Yin, Carreno y Zhang, 2021, y Parakongonema sínica Zhang, Yin, Carreno y Zhang, 2021 no fueron recuperadas por ambos análisis, por lo que no se logró resolver con claridad su relación con el resto de los histrignatidos a partir de los 2 métodos utilizados y la información disponible.
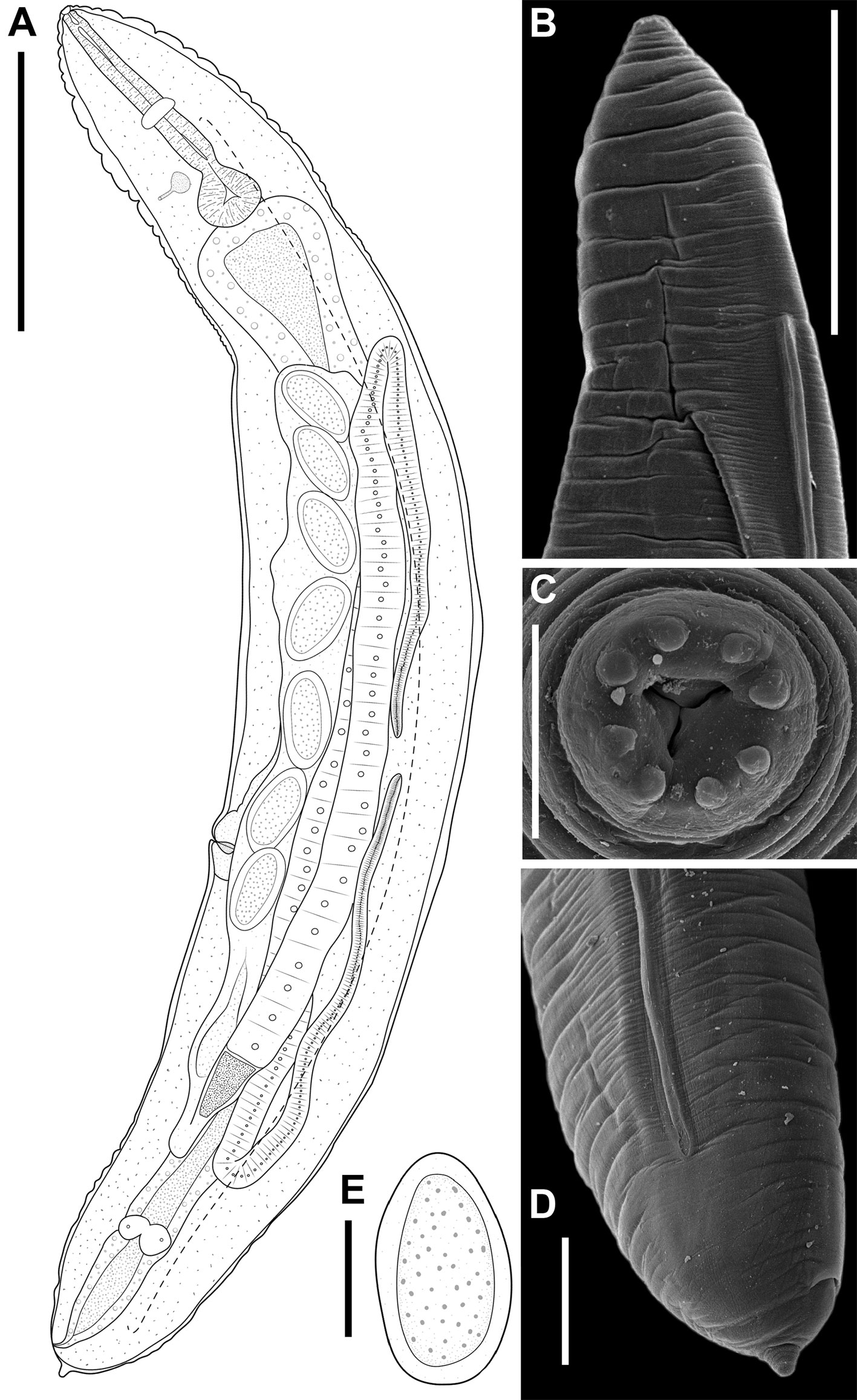
Tabla 2
Números de acceso de las secuencias depositadas en GenBank de los taxones de Oxyuridomorpha incluidos en el análisis filogenético.
| Especies | Familia | SSU | LSU | Hospedero |
| Oxyuroidea | ||||
| Spauligodon atlanticus | Oxyuridae | JF829230 | KJ778099 | Gallotia atlantica (Reptilia) |
| Ozolaimus linstowi | Oxyuridae | KJ632671 | KJ632667 | Iguana iguana (Reptilia) |
| Thelastomatoidea | ||||
| Leidynema appendiculata | Thelastomatidae | KY057035 | KC540759 | Periplaneta fuliginosa (Insecta) |
| Hammerschmidtiella diesingi | Thelastomatidae | KJ632670 | EU365628 | Blatta orientalis (Insecta) |
| Pseudonymus spirotheca | Pseudonymidae | KJ632669 | KF771649 | Hydrophilus piceus (Insecta) |
| Travassosinema claudiae | Travassosinematidae | KX844644 | KX844645 | Parafontaria laminata (Diplopoda) |
| Cephalobellus brevicaudatus | Thelastomatidae | MF668724 | MF668725 | Tipula sp. (Insecta) |
| Xyo pseudohystrix | Hystrignathidae | MK332329 | MK332328 | Odontotaenius disjunctus (Insecta) |
| Lepidonema magnum | Hystrignathidae | MH577324 | MH569782 | Passalus interstitialis (Insecta) |
| Lepidonema zuritai n. sp. | Hystrignathidae | OK576056 | OK575763 | Heliscus tropicus (Insecta) |
| Lepidonema zuritai n. sp. | Hystrignathidae | – | OK575766 | Heliscus tropicus (Insecta) |
| Coynema poeyi | Hystrignathidae | MH577322 | MH244509 | Passalus pertyi (Insecta) |
| Longior alius | Hystrignathidae | MH411158 | KX427527 | Passalus pertyi (Insecta) |
| Longior similis | Hystrignathidae | MH411157 | KX427531 | Passalus interstitialis (Insecta) |
| Longior lamothei | Hystrignathidae | – | MH411127 | Passalus punctiger (Insecta) |
| Longior zumpimito | Hystrignathidae | MH411155 | MH411128 | Passalus punctatostriatus (Insecta) |
| Longior surieli | Hystrignathidae | MH411154 | MH411126 | Passalus dominicanus (Insecta) |
| Hystrignathus rigidus | Hystrignathidae | MH411156 | MH411129 | Odontotaenius disjunctus (Insecta) |
| Urbanonema osorioi | Hystrignathidae | MN578050 | MN578048 | Verres hageni (Insecta) |
| Urbanonema osorioi | Hystrignathidae | MN578051 | MN578047 | Verres hageni (Insecta) |
| Urbanonema osorioi | Hystrignathidae | MN578052 | MN578046 | Verres hageni (Insecta) |
| Tuhmai garciaprietoi | Hystrignathidae | MN578053 | MN578049 | Vindex agnoscendus (Insecta) |
| Tuhmai garciaprietoi | Hystrignathidae | MT069968 | MT070420 | Vindex agnoscendus (Insecta) |
| Huntia morffei | Hystrignathidae | – | MZ330362 | Ceracupes fronticornis (Insecta) |
| Hystrignathus nunashae | Hystrignathidae | MW534554 | MW534555 | Passalus interruptus (Insecta) |
| Hystrignathus nunashae | Hystrignathidae | – | MW534556 | Passalus interruptus (Insecta) |
| Hystrignathus sp. | Hystrignathidae | – | GQ368469 | Passalidae gen. sp. (Insecta) |
| Phalacronema vegae n. sp. | Hystrignathidae | OK576057 | OK575764 | Passalus cognatus (Insecta) |
| Phalacronema vegae n. sp. | Hystrignathidae | OK576058 | OK575765 | Passalus cognatus (Insecta) |
| Klossnema viguerasi | Hystrignathidae | MW030189 | MW030185 | Passalus pertyi (Insecta) |
| Parahystrignathus tongbiguanensis | Hystrignathidae | – | MZ330365 | Ceracupes fronticornis (Insecta) |
| Parakongonema sinica | Hystrignathidae | – | MZ330359 | Aceraius grandis (Insecta) |
| Parakongonema sinica | Hystrignathidae | – | MZ330363 | Aceraius grandis (Insecta) |
| Coronostomatoidea | ||||
| Coronostoma claireae | Coronostomatidae | – | MH244505 | Narceus gordanus (Diplopoda) |
| Triumphalisnema zuuei | Traklosiidae | MH220046 | MN628599 | Proculejus hirtus (Insecta) |
| Triumphalisnema zuuei | Traklosiidae | MH220047 | MN628600 | Proculejus hirtus (Insecta) |
Por otro lado, algunos subclados bien sustentados por ambos métodos son aquellos conformados por Lepidonema magnum, Hystrignathus rigidus Leidy, 1850, Urbanonema osorioi Garduño-Montes de Oca y Oceguera-Figueroa 2020 y Hystrignathus nunashae Garduño-Montes de Oca, 2022, así como el del resto de las especies de Longior, y aquel formado por Lepidonema zuritai n. sp. y Xyo pseudohystrix. Este último clado resulta particularmente notorio a la luz de las afinidades geográficas y morfológicas entre ambas especies, a saber: la presencia de espinas cuticulares y la configuración del aparato reproductor, así como la relativa cercanía entre el sureste de EUA (donde se distribuye Xyo psudohystrix) y el noreste de México (donde se encuentra la localidad tipo de Lepidonema zuritai n. sp.). No obstante, aún son inciertas las relaciones entre estos clados, por lo que la adición de diferentes taxones, así como la inclusión de otro tipo de marcadores (ej. mitocondriales) podrían contribuir a tener una mayor claridad con respecto a las relaciones evolutivas entre los subclados de Hystrignathidae (fig. 3).
Discusión
Las especies de nemátodos asociadas con los escarabajos Passalidae incluidas en el presente estudio, forman un grupo monofilético apoyado por ambos métodos, este patrón sugiere cierto grado de especificidad en la interacción Hystrignathidae-Passalidae, tal patrón puede ser analizado a mayor detalle en futuros estudios que podrían permitir acercarse a las causas ecológicas o históricas de la aparente restricción hospedatoria, sugerida por la hipótesis filogenética obtenida en el presente trabajo. Adicionalmente, es conveniente considerar que la mayoría de las especies de nemátodos asociadas con Passalidae que se incluyen en nuestro análisis, se distribuyen en Norteamérica, con 4 especies de Hystrignathidae provenientes de Asia y 1 de Sudamérica. Por lo tanto, sería necesario ampliar el muestreo taxonómico para incluir diversas regiones, con la finalidad de distinguir entre posibles patrones de asociación geográfica y patrones de asociación hospedatoria. En resumen, expandir el muestreo geográfico y taxonómico, así como el diseño de estudios enfocados en entender la variación poblacional, podrían ser los primeros pasos para comprender cuáles son los factores ecológicos y evolutivos involucrados en la diversificación de Hystrignathidae.
Agradecimientos
A Milan Janda y a Oscar Pérez-Flores por el trabajo de campo; a los entomólogos Viridiana Vega-Badillo y Edwin Ariza Marín (INECOL) por la identificación de los hospederos. A Andrea Rebollo, Andrea Jiménez, Laura Márquez-Valdelamar y Nelly María López-Ortiz por su asistencia en el laboratorio de Biología Molecular. A Alejandro Oceguera-Figueroa por su imprescindible apoyo en este y otros estudios. Este proyecto fue financiado por el Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica IN215722 (PAPIIT-UNAM) y el Consejo Nacional de Ciencia y Tecnología (Conacyt) Ciencia Básica 220408, a cargo de Alejandro Oceguera Figueroa. Agradezco al Conacyt la beca nacional otorgada para la realización del programa de doctorado en el Posgrado en Ciencias Biológicas, UNAM. Al Posgrado en Ciencias Biológicas, UNAM. También agradezco a Berenit Mendoza-Garfias por su asistencia en el procesamiento de micrografías en LANABIO IB-UNAM, y a Luis García Prieto por su asistencia en la incorporación del material helmintológico en la CNHE-IBUNAM. Finalmente, agradezco a la Willi Hennig Society por subsidiar el programa TNT de libre acceso.
Referencias
Adamson, M. L. y Van Waerebeke, D. (1992). Revision of the Thelastomatoidea, Oxyurida of invertebrate hosts III. Hystrignathidae. Systematic Parasitology, 22, 111–130. https://doi.org/10.1007/BF00009604
Blaxter, M. L., De Ley, P., Garey, J. R., Liu, L. X., Scheldeman, P., Vierstraete, A. et al. (1998). A molecular evolutionary framework for the phylum Nematoda. Nature, 392, 71–75. https://doi.org/10.1038/32160
Carreno, R. A. (2014). The systematics and evolution of pinworms (Nematoda: Oxyurida: Thelastomatoidea) from invertebrates. The Journal of parasitology, 100, 553–560. https://doi.org/10.1645/14-529.1
Clark, W. C. (1978). Nematoda (Oxyuroidea: Thelastomatidae) from Australian passalid beetles (Coleoptera: Passalidae) with descriptions of new species. Australian Journal of Zoology, 26, 603–615.
Cobb, N. A. (1898). Extract from MS report on the parasites of stock. The Agricultural Gazette of New South Wales, 9, 296–321.
De Man, J. G. (1884). Die frei in der reinen Erde und im süssen Wasser lebenden Nematoden der Niederländischen Fauna: eine systematisch-faunistische Monographie. Leiden: E.J. Brill. https://doi.org/10.5962/bhl.title.46884
García, N., Ventosa, M. L. y Morffe, J. (2009). Dos especies nuevas de los géneros Lepidonema y Longior (Thelastomatoidea: Hystrignathidae) de la Isla de la Juventud, Cuba. Solenodon, 8, 1–7.
Garduño-Montes de Oca, E. U., Vega-Badillo, V., López-Caballero, J., Ramírez-Cañas, S. A. y Mata-López, R. (2020). A new species of Triumphalisnema (Nematoda: Coronostomatoidea) parasite of Proculejus hirtus (Coleoptera: Passalidae) from Mexico, and the position of the family in the Oxyuridomorpha. The Journal of Parasitology, 106, 172–179. https://doi.org/10.1645/19-64.
Goloboff, P. A., Farris, J. S. y Nixon, K. C. (2008). TNT, a free program for phylogenetic analysis. Cladistics, 24, 774–786. https://doi.org/10.1111/j.1096-0031.2008.00217.x.
Hodda, M. (2007). Phylum Nematoda. Zootaxa, 1668, 265–293. https://doi.org/10.11646/zootaxa.1668.1.14
Holterman, M., Van der Wurff, A., Van den Elsen, S., Van Megen, H., Bongers, T., Holovachov, O. et al. (2006). Phylum-wide analysis of SSU rDNA reveals deep phylogenetic relationships among nematodes and accelerated evolution toward crown clades. Molecular Biology and Evolution, 23, 1792–1800. https://doi.org/10.1093/molbev/msl044
Katoh, K., Rozewicki, J. y Yamada, K. D. (2019). MAFFT online service: Multiple sequence alignment, interactive sequence choice and visualization. Briefings in Bioinformatics, 20, 1160–1166. https://doi.org/10.1093/bib/bbx108
Kearse, M., Moir, R., Wilson, A., Stones-Havas, S., Cheung, M., Sturrock, S. et al. (2012). Geneious Basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data. Bioinformatics, 28, 1647–1649. https://doi.org/10.1093/bioinformatics/bts199
Maddison, W. P. y Maddison, D. R. (2019). Mesquite: a modular system for evolutionary analysis. Versión 3.04. Última consulta el 27 enero, 2022 de: http://mesquiteproject.org
Medlin, L., Elwood, H. J., Stickel, S. y Sogin, M. L. (1988). The characterization of enzymatically amplified eukaryotic 16S-like rRNA-coding regions. Gene, 71, 491–499. https://doi.org/10.1016/0378-1119(88)90066-2
Morffe, J. y García, N. (2010). Two new species of nematode (Oxyurida, Hystrignathidae) parasites of Passalus interstitialis Escholtz, 1829 (Coleoptera, Passalidae) from Cuba and a new locality for Longior similis Morffe, García and Ventosa, 2009. Zookeys, 49, 1–11. https://doi.org/10.3897/zookeys.49.402
Morffe, J., García, N., Hasegawa, K. y Breugelmans, K. (2021). Morphological and molecular characterization of Klossnema viguerasi n. sp. (Nematoda: Oxyuridomorpha: Hystrignathidae) from a Cuban passalid beetle (Coleoptera: Passalidae), first record of the genus for Cuba. Zootaxa, 4933, 391–402. https://doi.org/10.11646/zootaxa.4933.3.6
Nunn, G. B. (1992). Nematode molecular evolution: an investigation of evolutionary patterns among nematodes based upon DNA sequences (Tesis doctoral). University of Nottingham, Nottingham, Reino Unido.
Posada, D. y Crandall, K. A. (1998). MODELTEST. Testing the model of DNA substitution. Bioinformatics, 14, 817–818. https://doi.org/10.1093/bioinformatics/14.9.817
Rambaut, A. (2006). FigTree Tree figure drawing tool, version 1.4. 2. 2016. Última consulta el 19 enero, 2022: http://tree.bio.ed.ac.uk/software/figtree/
Rambaut, A., Drummond, A. J., Xie, D., Baele, G. y Suchard, M. A. (2018). Posterior summarization in Bayesian phylogenetics using Tracer 1.7. Systematic Biology, 67, 901–904. https://doi.org/10.1093/sysbio/syy032
Reyes-Castillo, P. (1970). Coleoptera, Passalidae: Morphology and division into large groups; American genera. Folia Entomológica Mexicana, 20, 217–232.
Reyes-Castillo, P. (2000). Coleoptera Passalidae de México. En F. Martín-Piera, J. J. Morrone y A. Melic (Eds.), Hacia un proyecto CYTED para el inventario y estimación de la diversidad entomológica en Iberoamérica (pp. 171–182). Zaragoza, España: Sociedad Entomológica Aragonesa.
Ronquist, F., Teslenko, M., Van Der Mark, P., Ayres, D. L., Darling, A., Höhna, S. et al. (2012). MrBayes 3.2. Efficient Bayesian phylogenetic inference and model choice across a large model space. Systematic Biology, 61, 539–542. https://doi.org/10.1093/sysbio/sys029
Zhang, N., Yin, S., Carreno, R. A. y Zhang, L. (2021). Three new genera and new species of hystrignathid nematodes (Nematoda: Thelastomatoidea) from passalid beetles (Insecta: Passalidae) from Yunnan Province, China with phylogenetic analysis of Hystrignathidae. Zootaxa, 5071, 206–222. https://doi.org/10.11646/zootaxa.5071.2.2




