La Ecología Evolutiva del Desarrollo en México
Elena R. Álvarez-Buylla(a)(b), Adriana Garay-Arroyo(a)(b), Berenice García-Ponce de León(a)(b), María de la Paz Sánchez(a)(b), Emmanuel González-Ortega(a)(b), José Dávila-Velderrain(a)(b)(c), Juan Carlos Martínez-García(a)(b)(d), Alma Piñeyro-Nelson(b)(e)⁎ ✉
Resumen
La biología evolutiva se enfoca al estudio de los patrones de variación fenotípica heredables dentro de las poblaciones y su dinámica en tiempos transgeneracionales. Históricamente, los modelos evolutivos a nivel de las poblaciones se han desarrollado bajo supuestos simples. Dos de ellos particularmente importantes son: 1) el cambio genético es un indicador directo de la variación fenotípica y 2) existe aditividad de los impactos genéticos sobre el fenotipo. Un modelo más certero de evolución biológica debería de considerar explícitamente el mapeo entre genotipo y fenotipo y viceversa, ya que los mecanismos que median el desarrollo de los organismos y su interacción con el ambiente son los que generan la variación fenotípica. Una perspectiva dinámica no lineal es imprescindible para entender cómo se genera la variación fenotípica dado un fondo genético particular. Dicho de otra manera, es necesario entender los mecanismos sistémicos del mapeo entre genotipo y fenotipo en un contexto ecológico y evolutivo. El campo que comprende este enfoque es el de la Ecología Evolutiva del Desarrollo (Eco-Evo-Devo), el cual tiene además un enfoque sistémico. En México este campo ha crecido de manera incipiente a través del análisis de los mecanismos de desarrollo con enfoques de biología de sistemas, principalmente. En este artículo resumimos avances de este campo de estudio en México y cuáles son los principales grupos de investigación en el país, tanto en plantas como en animales. También evaluamos los impactos biomédicos de tomar esta perspectiva.
(http://creativecommons.org/licenses/by-nc-nd/4.0/).
Palabras clave:
Eco-Evo-Devo; Arabidopsis; Epigenética; Modelos de redes complejas; Mecanismos del desarrollo
Developmental Evolutionary Ecology in Mexico
Abstract
Evolutionary biology is largely concerned with the patterns of heritable phenotypic variation within populations and its dynamics during long transgenerational time periods. Historically, population-level models in evolution have been developed under certain simplifying assumptions. Two salient assumptions are: 1) the idea of genetic change as a direct indicator of phenotypic variation, and 2) the additivity of genetic effects on the phenotype. A more faithful model of biological evolution should explicitly consider a genotype-phenotype map and back mediated by a developmental mechanism, which specifies how phenotypic variation is generated in different environments, in an analogous way to that in which positional information emerges as a result of the feedback between internal and external restrictions. A dynamic non-linear perspective is thus mandatory to understand how phenotypic variation is generated given a genetic background; or in other words, to study the mechanistic basis of the genotype-phenotype map within an ecological and evolutionary context. The field that focuses on this is Ecological Evolutionary Developmental Biology (Eco-Evo-Devo) with a systemic approach and, in Mexico, it is an emergent field. In this paper we summarize the main Eco-Evo-Devo contributions with such approach from Mexican laboratories using plants and animals. We also discuss the biomedical implications of such an approach.
Keywords:
Eco-Evo-Devo; Arabidopsis; Epigenetics; Complex network models; Developmental mechanisms;
Introducción
¿Qué es la Ecología Evolutiva del Desarrollo?
En la síntesis moderna de la teoría de la evolución se integró a la teoría darwiniana de evolución por selección natural las leyes mendelianas de la herencia y la genética de poblaciones ( Dobzhansky, 1937; Fisher, 1930; Haldane, 1932; Wright, 1931 ). Desde entonces, el estudio de la evolución se ha enfocado, preponderantemente, a entender cómo las fuerzas evolutivas y los sistemas reproductivos actúan sobre variantes alélicas o genotípicas en distintos ambientes ( Dobzhansky, 1937 ). Los modelos de evolución implicados en esta síntesis asumen un simple mapeo del genotipo al fenotipo y efectos generalmente aditivos de los distintos genes sobre los fenotipos. En la síntesis moderna, sin embargo, se dejó fuera la biología del desarrollo, campo que estudia el conjunto de procesos que justamente están implicados en el mapeo complejo y dinámico de la variación genotípica a la fenotípica, incluyendo el papel del ambiente en este mapeo.
La biología Evolutiva del Desarrollo (Evo-Devo) se ha enfocado al estudio de los mecanismos que rigen el desarrollo de los organismos (de aquí en adelante denominado simplemente «desarrollo») desde la fecundación hasta la senectud, y cómo estos mecanismos afectan los patrones de especiación (macroevolución) y la evolución a nivel de las poblaciones (microevolución). Además, al estudiar la evolución de los propios mecanismos de desarrollo, la Evo-Devo ayuda a explicar patrones de cambio fenotípico entre especies a distintos niveles filogenéticos. Por otro lado, la Ecología Evolutiva del Desarrollo (Eco-Evo-Devo), incorpora la Evo-Devo a la ecología con el fin de explorar el papel de factores ecológicos en el desarrollo y la emergencia de nuevos rasgos fenotípicos, la microevolución de estos rasgos y la fijación de ciertos fenotipos dentro de las especies o en niveles filogenéticos superiores. También explora cómo emergen la plasticidad fenotípica (i.e., variación fenotípica de un mismo genotipo en distintos ambientes) y las normas de reacción de un genotipo (i.e., patrón de cambio fenotípico de un genotipo ante variaciones de un factor ambiental). Finalmente, se investiga cuál es el papel de la plasticidad en la evolución fenotípica adaptativa y no adaptativa ( Espinosa-Soto, 2016; Espinosa-Soto, Martin y Wagner, 2011a, b ).
Recientemente se ha acumulado evidencia de que el ambiente puede tener un papel importante en la emergencia de nuevos fenotipos y su evolución. Se ha visto que, en muchos casos, esto sucede a través de cambios epigenéticos. Al igual que la variación genética, la variación epigenética (i.e., variación de factores no genéticos producidos por cambios en el ambiente celular) se puede heredar de generación en generación, impactando a la evolución fenotípica, e incluso fijándose en ciertos linajes o especies ( Gilbert y Epel, 2009; Sánchez et al., 2015 ). En resumen, la integración de la ecología, de la biología del desarrollo y de la evolución en el campo de estudio de la Eco-Evo-Devo rompe con las concepciones causa-efecto unidireccionales y simples de las visiones genocéntricas, y plantea una nueva síntesis para la biología evolutiva ( Gilbert, 2012; figs. 1 y 2).
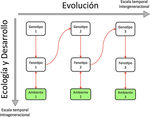
Figura 1
Ecología Evolutiva del Desarrollo. El desarrollo de los organismos y su retroalimentación con el ambiente (ecología) ocurren en tiempos intrageneracionales, desde la fertilización, el desarrollo embrionario y las diferentes fases del ciclo de vida subsecuentes. Por otro lado, la evolución opera a nivel de los genotipos, de los mecanismos de desarrollo y de los fenotipos en tiempos intergeneracionales. Esta última escala temporal implica cambios genéticos transgeneracionales o incluso la fijación de rasgos (y mecanismos de desarrollo subyacentes) en algunos linajes. Modificado de Lewontin, 1974).
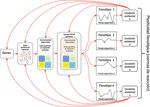
Figura 2
Ecología del desarrollo. Esquema que resume el enfoque de modelos emergentes en los que se usan modelos dinámicos de redes moleculares acopladas por campos físico-químicos y de paisajes epigenéticos para estudiar el desarrollo (la diferenciación celular y la morfogénesis). Estos modelos permiten indagar la emergencia de fenotipos (a distintos niveles de organización) en retroalimentación con el ambiente, en la búsqueda de explicar la plasticidad y las normas de reacción. Los paisajes epigenéticos pueden alterarse por factores ambientales y esto puede traducirse en un cambio fenotípico que explica la plasticidad.
En México se han estudiado temas de la biología del desarrollo y de la evolución del desarrollo con enfoques de sistemas complejos con el fin de investigar los mecanismos sistémicos de la diferenciación celular y la morfogénesis, tratando de entender el origen de los fenotipos, su variación y evolución ( Mendoza y Álvarez-Buylla, 1998, 2000; Mendoza, Thieffry y Álvarez-Buylla, 1999; Espinosa-Soto, Immink, Angenent, Álvarez-Buylla y de Folter, 2014; Espinosa-Soto et al., 2011a, b; Vergara-Silva, 2003; Vergara-Silva, Martínez-Castilla y Álvarez-Buylla, 2000 , y revisiones en Álvarez-Buylla et al., 2007; Álvarez-Buylla et al., 2007; Álvarez-Buylla, Azpeitia, Barrio, Benítez y Padilla-Longoria, 2010; Álvarez-Buylla et al., 2010; Álvarez-Buylla, Dávila-Velderrain y Martínez-García, 2016; Dávila-Velderrain, Martínez-García y Álvarez-Buylla, 2016; Garay-Arroyo, Sánchez, García-Ponce, Azpeitia y Álvarez-Buylla, 2012 ). Recientemente, se han iniciado investigaciones en torno a la biología ecológica y evolutiva del desarrollo. En particular, algunos de los autores del presente artículo han estudiado las consecuencias fenotípicas de la retroalimentación entre mecanismos genéticos y epigenéticos con la ecología de los organismos y sus consecuencias para la evolución fenotípica ( Álvarez-Buylla et al., 2007; Álvarez-Buylla et al., 2007; Álvarez-Buylla et al., 2008, 2013, 2016; Chaos et al., 2006; Espinosa-Soto, Padilla-Longoria y Álvarez-Buylla, 2004 ). Otros grupos en México han abordado enfoques bioinformáticos y experimentales que son relevantes para el estudio Eco-Evo-Devo en plantas ( Rosas et al., 2013 ), animales (e.g., Santacruz, Tinoco-Cuellar, Rangel-Huerta y Maldonado, 2015 ) y en desarrollo de exploraciones teóricas ( Espinosa-Soto, 2016; Espinosa-Soto et al., 2011a, b ). A continuación presentamos los avances en varios temas abordados en México a través de la Eco-Evo-Devo.
Modelos dinámicos de redes de la diferenciación celular y morfogénesis: hacia un enfoque sistémico y mecanístico de la Eco-Evo-Devo
A nivel del desarrollo de los organismos y su interacción con el ambiente emerge un tipo de regulación epigenética de las redes de interacción de componentes genéticos y no genéticos que operan a nivel de células, tejidos, órganos y organismos ( fig. 2 ). Dichas redes interactúan de manera no lineal y se retroalimentan entre sí a través de sus componentes. Las redes se pueden articular con vías de transducción de señales internas y ambientales, con fuerzas físicas y sustancias químicas que establecen campos micro- y mesoscópicos con el ambiente ( Álvarez-Buylla et al., 2016; Barrio et al., 2013; Pérez-Ruiz et al., 2015 ; fig. 1).
Para organismos multicelulares, se ha planteado que las redes de interacción que producen distintas configuraciones genéticas estables o de actividad de proteínas, conocidas como redes metaestables, subyacen a la diferenciación celular (e.g., Dávila-Velderrain, Villarreal y Álvarez-Buylla, 2015 ). Esta propuesta conceptual, originalmente desarrollada por Stuart Kauffman para redes metabólicas aleatorias ( Kauffman, 1969 ), se validó por primera vez en México con una red genética regulatoria de la diferenciación celular del desarrollo floral en Arabidopsis thaliana (Arabidopsis de aquí en adelante) ( Álvarez-Buylla et al., 2016; Chaos et al., 2006; Dávila-Velderrain y Álvarez-Buylla, 2014; Espinosa-Soto et al., 2004; Mendoza y Álvarez-Buylla, 1998, 2000; Mendoza et al., 1999 ), una especie de la familia Brassicaceae. Posteriormente, se simuló la emergencia de patrones de transiciones celulares, ordenados en el tiempo, usando modelos estocásticos ( Álvarez-Buylla et al., 2008 , ver también sección siguiente). Más tarde, se modeló la interacción de redes que intervienen en la diferenciación de células únicas (e.g., Azpeitia, Benítez, Vega, Villarreal y Álvarez-Buylla, 2010; Barrio, Hernández-Machado, Varez, Romero-Arias y Álvarez-Buylla, 2010; Benítez, Espinosa-Soto, Padilla-Longoria y Álvarez-Buylla, 2008; Benítez, Espinosa-Soto, Padilla-Longoria, Díaz y Álvarez-Buylla, 2007 ). Por otro lado, se han propuesto modelos de dinámicas cooperativas (e.g., proliferación celular, campos físicos y gradientes químicos) que muestran que aún sin la consideración explícita de las redes regulatorias transcripcionales, se recuperan patrones celulares y morfogenéticos similares a los observados en los seres vivos. En estos trabajos ( Barrio et al., 2010, 2013; Benítez et al., 2008; Hernández-Hernández, Rueda, Caballero, Álvarez-Buylla y Benítez, 2014 ) se postula un posible mecanismo sobre el origen de la información posicional que guía la diferenciación celular y los patrones espacio-temporales de tal diferenciación. Todos estos estudios sugieren que la información posicional no es meramente intracelular o extracelular (de posición) como ha sido el planteamiento clásico en la biología del desarrollo ( Wolpert, Tickle y Martínez-Arias, 2015 ), sino una propiedad emergente de la retroalimentación entre las redes intracelulares y los campos físico-químicos o restricciones geométricas ( Álvarez-Buylla, Benítez y Espinosa-Soto, 2011; Álvarez-Buylla et al., 2016 ).
Modelos del paisaje epigenético: un marco teórico para la Eco-Evo-Devo
El modelo de paisaje epigenético (PE), propuesto originalmente por Waddington (1957) , es un marco conceptual que vincula a la genética, la biología del desarrollo y la evolución. La evolución de la variación fenotípica involucra múltiples escalas temporales y contempla restricciones inherentes a la naturaleza física de los sistemas biológicos. Estos aspectos han dificultado desentrañar los mecanismos que originan y dirigen la variación fenotípica, cuyo entendimiento es central para comprender como actúa la selección natural en las poblaciones. Waddington (1957) , de manera visionaria, integró implícitamente estas dificultades al proponer un modelo intuitivo y simple al imaginar el proceso de desarrollo de un organismo de manera análoga al movimiento de una pelota que rueda sobre un paisaje rugoso. En el PE, la pelota pasa por valles y crestas que representan los estados fenotípicos de un organismo, determinando o favoreciendo ciertas trayectorias de desarrollo sobre otras. La parte más ingeniosa del modelo de PE reside en la propuesta mecanística para explicar cómo emerge la forma del paisaje y cómo cambia esta forma en tiempos evolutivos. El componente genético del modelo original del PE radica en la consideración de una red compleja de interacciones de genes que subyace al paisaje y determina de manera importante su topografía. Por lo tanto, las transiciones fenotípicas ordenadas en tiempo y espacio, que ocurren durante el proceso de desarrollo, son de alguna manera reguladas por estas redes. Durante el proceso de evolución las modificaciones genéticas alteran la red subyacente y, a su vez, la forma del paisaje. A partir de los modelos de redes genéticas referidos en el apartado anterior, es posible modelar el PE ( Álvarez-Buylla et al., 2008 ). Aunque no todos los cambios del PE producirán un efecto fenotípico observable y sujeto a selección. Para Waddington, el papel de la selección natural radica en inducir la canalización del desarrollo y favorecer aquellas vías de desarrollo en el PE que, de manera consistente, den lugar a la formación de un organismo adulto, bajo las condiciones ambientales en las que el organismo normalmente se desarrolla ( Waddington, 1953 ). Este mismo proceso implica también que cambios drásticos en el ambiente pueden alterar el paisaje, y por lo tanto, producir variación fenotípica. Además, los cambios genéticos pueden interactuar con el ambiente y eventualmente se pueden fijar nuevas trayectorias o vías de desarrollo dando lugar a variación fenotípica novedosa que puede ser heredable ( Waddington, 1942, 1953; Wilkins, 2008 ).
En México, el estudio de PE se inició dentro del marco teórico de las redes de regulación genéticas, en particular para el caso de la red de determinación de órganos florales en plantas del género Arabidopsis (Álvarez-Buylla et al., 2008 ) y más recientemente se ha avanzado en propuestas teóricas ( Dávila-Velderrain, Juárez-Ramiro, Martínez-García y Álvarez-Buylla, 2015; Dávila-Velderrain, Martínez-García y Álvarez-Buylla, 2015a, b; Villarreal, Padilla-Longoria y Álvarez-Buylla, 2012 ). Las redes regulatorias se han establecido como modelos concretos para el estudio de los mecanismos subyacentes a los procesos de diferenciación celular y de morfogénesis durante el desarrollo, por un lado, y de unidades funcionales que pueden estar sujetas a la selección natural, por otro ( Espinosa-Soto et al., 2004; Mendoza y Álvarez-Buylla, 1998 ). Esta última propuesta fue verificada empíricamente a través del análisis genómico de la conservación de la red regulatoria del desarrollo floral en diferentes angiospermas ( Dávila-Velderrain, Servin-Márquez y Álvarez-Buylla, 2013 ). También se han realizado contribuciones importantes sobre el papel de la plasticidad fenotípica (y la variabilidad no genética en general) en la adaptación y consistencia de sistemas biológicos en el contexto de la evolución de la modularidad de las plantas ( Espinosa-Soto, 2016; Espinosa-Soto y Wagner, 2010; Espinosa-Soto et al., 2011a, b ). Estos últimos estudios han generado predicciones interesantes sobre el papel que puede desempeñar la plasticidad de los genotipos en la evolución adaptativa ( Espinosa-Soto et al., 2011a, b ). Dentro de la misma área teórico/computacional, en el Centro de Ciencias Físicas de la Universidad Nacional Autónoma de México (UNAM) se ha estudiado extensamente la evolución de las propiedades genéricas de redes regulatorias ( Aldana, Balleza, Kauffman y Resendiz, 2007; Balleza et al., 2008; Torres-Sosa, Huang y Aldana, 2012 ) y el papel de la variabilidad no genética y su función en la adaptación ( Motta, Cluzel y Aldana, 2015).
En el futuro, sería muy interesante contar en México con un sistema experimental que permitiera validar los modelos teóricos de redes y de PE en contextos ecológicos y evolutivos particulares. Por ahora, se ha avanzado en estudios comparativos de genes que son claves en módulos de desarrollo ya identificados y su regulación epigenética, así como el impacto de estos módulos en la morfología de las especies bajo estudio ( Álvarez-Buylla et al., 2000; Álvarez-Buylla et al., 2000; Ambrose et al., 2006; Espinosa-Matías, Vergara-Silva, Vázquez-Santana, Martínez-Zurita y Márquez-Guzmán, 2012; Rodríguez-Mega et al., 2015; Vázquez-Lobo et al., 2007; Vergara-Silva et al., 2000, 2003; Yockteng, Almeida, Morioka, Álvarez-Buylla y Specht, 2013 ). También se han desarrollado estudios filogenéticos y de evolución molecular evaluando el papel de la selección natural a nivel de secuencias de genes y proteínas para familias de genes reguladores importantes durante el desarrollo ( Álvarez-Buylla et al., 2000; Hernández-Hernández, Martínez-Castilla y Álvarez-Buylla, 2007; Martínez-Castilla y Álvarez-Buylla, 2003 ). Se resumen a continuación los avances más concretos en la Eco-Evo-Devo de plantas y animales en México
Estudios de Eco-Evo-Devo en plantas
El uso de diferentes ecotipos de Arabidopsis , así como especies emparentadas, ha resultado particularmente útil como sistema modelo para los estudios Eco-Evo-Devo de plantas en México. A partir de los estudios de la red regulatoria de especificación de los órganos de la flor (sépalos, pétalos, estambres y carpelos) de esta planta, en el país se han propuesto módulos reguladores involucrados en procesos de diferenciación celular y morfogénesis, incluyendo: la transición del estado vegetativo al de floración ( Pérez-Ruiz et al., 2015 ), el establecimiento y mantenimiento del patrón celular en el nicho de células troncales de la raíz ( Azpeitia et al., 2010 ) y la subdiferenciación celular en la epidermis de la hoja y la raíz ( Benítez et al., 2007, 2008 ). Algunos de estos procesos de desarrollo son particularmente importantes para estudios de Eco-Evo-Devo, aunque será necesario extender este tipo de enfoques a otras especies. Aquí resumimos los avances en algunos de los sistemas estudiados en México.
Mecanismos de regulación epigenética: relevancia para la Eco-Evo-Devo
El fenotipo de un organismo está determinado tanto por su material genético como por factores epigenéticos. Estos últimos pueden modificarse en función de la interacción del organismo con los factores ambientales, bióticos y abióticos, que afectan la embriogénesis y el desarrollo postembrionario. Los mecanismos epigenéticos alteran la expresión génica mediante la remodelación de la cromatina sin alterar la secuencia de ADN; estas alteraciones, que resultan de la interacción de mecanismos moleculares de la cromatina con factores ambientales, pueden ser heredables entre generaciones ( Berger, Kouzarides, Shiekhattar y Shilatifard, 2009; Richards, 2006 ). El carácter hereditario de algunas variaciones epigenéticas demanda un replanteamiento del concepto de herencia biológica y sus implicaciones evolutivas ( Jablonka y Lamb, 2005 ). La regulación epigenética modula respuestas fisiológicas y del desarrollo de los organismos ante cambios en el ambiente. Por ejemplo, en las plantas tal regulación se da en los mecanismos de defensa contra herbívoros, así como en las respuestas plásticas de los organismos ante diferentes situaciones de estrés ( Feng, Jacobsen y Reik, 2010; Sánchez et al., 2015; Schuettengruber, Martínez, Iovino y Cavalli, 2011 ). Los fenómenos epigenéticos en los seres humanos tienen implicaciones directas sobre enfermedades hereditarias o somáticas, tales como varios procesos de cáncer y mecanismos de defensa contra patógenos ( Egger, Liang, Aparicio y Jones, 2004 ). Además, al ser mecanismos que pueden integrar las señales ambientales, tienen implicaciones directas en campos como la agroecología.
En el Laboratorio de Ecología Molecular y Epigenética del Instituto de Ecología (UNAM) se estudian los mecanismos epigenéticos involucrados en diferentes procesos del desarrollo de las plantas y su interacción con el medio ambiente. Uno de estos mecanismos integra las señales ambientales durante varios procesos del desarrollo de las plantas y son controlados por 2 grupos de proteínas, uno de inhibición llamado complejo «Polycom» o PcG y uno de activación llamado complejo «Trithorax» o TrxG ( Schuettengruber et al., 2011 ). Estos complejos regulan procesos como la floración o la latencia de las semillas ( Sánchez et al., 2015; Song, Angel, Howard y Dean, 2012 ), el conocimiento de los mecanismos de acción de PcG y TrxG en el desarrollo de las plantas, y su interacción con factores ambientales, está contribuyendo a entender cómo las plantas pueden adaptarse a diferentes entornos ecológicos. El estudio de estos mecanismos también contribuye a entender la generación de variabilidad fenotípica heredable y reversible ya que esta variabilidad no depende de cambios producidos por mutaciones genéticas. Las implicaciones evolutivas de la epigénesis representan un campo reciente de investigación.
Respuestas plásticas a factores ambientales: transición a la floración
La transición del estado vegetativo al reproductivo (o simplemente floración) es un proceso en el desarrollo de las plantas cuya regulación tiene repercusiones directas en la probabilidad de dejar descendencia genéticamente diversa (a través de la producción de semillas) y, por lo tanto, incide directamente en la adecuación de estos organismos. La variación natural en los mecanismos que subyacen a este proceso ha permitido a las angiospermas adaptarse a una gran variedad de climas y latitudes, induciendo la floración en la época más propicia del año para la fecundación. Ya que la floración presenta respuestas plásticas a cambios ambientales, este proceso representa un sistema ideal para los estudios de Eco-Evo-Devo. Además, el movimiento de plantas por los humanos a regiones donde normalmente no crecían y las actividades de domesticación son ejemplos de fenómenos que han generado nuevas fuerzas selectivas que han cambiado los patrones temporales de floración de las plantas, constituyendo un caso de estudio particularmente interesante ( Nakamichi, 2015).
La floración involucra la dinámica de una red de regulación genética en la que participan más de 100 genes ( Koornneef, Alonso-Blanco y Vreugdenhil, 2004 ). Los estudios en Arabidopsis en México han permitido un avance considerable en el entendimiento de los mecanismos genético-moleculares que, en interacción con vías hormonales y factores ambientales, están involucrados en la regulación de la floración. Así, en el Laboratorio de Genética Molecular, Epigenética, Desarrollo y Evolución de Plantas del Instituto de Ecología (UNAM) se estudia el papel de genes «de caja» MADS (o MADS-box genes) en las redes de regulación genética que determinan el proceso de floración en Arabidopsis mediante una aproximación de genética reversa. Esta aproximación implica crecer plantas de Arabidopsis tipo silvestre y mutantes que florecen tardíamente bajo diferentes condiciones ambientales y en presencia/ausencia de hormonas inductoras de la floración ( Han, García-Ponce, Fonseca-Salazar, Álvarez-Buylla y Yu, 2008; Pérez-Ruiz et al., 2015; Tapia-López et al., 2008 ). Además, se ha estudiado la plasticidad fenotípica relacionada con genes MADS-box que regulan la floración a través de modelos de redes dinámicas y paisajes epigenéticos para explicar los patrones temporales de la floración ( Pérez-Ruiz et al., 2015 ). En el futuro se ampliarán estas investigaciones para estudiar floración en plantas de importancia agrícola como maíz, frijol o jitomate. Estas plantas son buenos sistemas de estudio ya que existe un gran vacío en el estudio Eco-Devo-Evo de plantas de regiones subtropicales y tropicales, donde el cambio del fotoperiodo es mínimo y no se experimentan cambios de temperatura drásticos a lo largo del año.
Se han llevado a cabo estudios Eco-Evo-Devo sobre la floración del maíz y sus parientes silvestres (teocintles). La floración de ciertas especies de teocintle es muy similar a la del maíz, en los aspectos morfológicos y fisiológicos, sin embargo, la floración en teocintle se inhibe cuando estas plantas crecen bajo un fotoperiodo de día largo. En contraste, algunas variedades de maíz pueden florecer independientemente del fotoperiodo. Esto se debe a que durante la domesticación del maíz se seleccionaron mutaciones en un gen regulador (denominado ZmCCT ) de la respuesta al fotoperiodo, pero aún se desconoce qué señales ambientales son las que disparan la floración en ambas especies ( Nakamichi, 2015 ). Por lo tanto, será importante indagar los genes involucrados en los cambios de la floración del teocintle al maíz. Este tema de investigación es particularmente importante para México, considerando que en el país existen miles de variedades de maíz con tiempos de transición a la floración contrastantes, mientras que cuenta con la mayoría de las subespecies y especies de teocintles.
Plasticidad del desarrollo de la raíz en respuesta a condiciones de estrés hiperosmótico
El déficit hídrico es uno de los factores ambientales que más alteran el desarrollo de las plantas ( Comstock, 2002; Davies y Zhang, 1991 ). Bajo condiciones de estrés hídrico, el crecimiento disminuye de manera proporcional a la severidad y a la magnitud de la condición de estrés y, si el estrés no es letal y se mantiene estable por un tiempo, el crecimiento de la planta se puede recuperar ( Chazen y Neumann, 1994; Skirycz et al., 2011; Veselov et al., 2002 ). Este tema de investigación está siendo abordado en el Instituto de Ecología de la UNAM estudiando la plasticidad del desarrollo de la raíz, y el papel de componentes genéticos y ambientales en tal plasticidad, en plantas de Arabidopsis . Se ha encontrado que el comportamiento celular a lo largo del eje longitudinal de la raíz y el desarrollo de la raíz está influenciado por hormonas, en particular por un gradiente de concentración de auxinas ( Garay-Arroyo et al., 2013 ). Sin embargo, la concentración de auxinas no explica todo este comportamiento celular y es necesario tomar en cuenta las dinámicas cooperativas que se establecen entre las concentraciones de auxinas, las tasas de proliferación celular y los cambios en las propiedades mecánico-elásticas de las células ( Barrio et al., 2013; Dávila-Velderrain et al., 2015; García-Cruz et al., 2016; Hernández-Hernández et al., 2014; Vieten et al., 2005 ). A pesar de que el desarrollo de la raíz es muy plástico y claramente está influenciado por factores ambientales, existe escasa información de los mecanismos moleculares que están involucrados en esta respuesta plástica. Específicamente, no se sabe cómo este órgano responde a condiciones de estrés osmótico por lo que es fundamental entender los mecanismos moleculares que subyacen a esa respuesta para ubicar el papel de factores genéticos y no genéticos en las respuestas plásticas al estrés ambiental, así como su evolución.
Otros investigadores en México están estudiando las bases moleculares de las respuestas al estrés hídrico en Arabidopsis y en plantas de frijol ( Contreras-Cubas, Palomar, Arteaga-Vázquez, Reyes y Covarrubias, 2012; Cuevas-Velázquez, Rendón-Luna y Covarrubias, 2014; Cuevas-Velázquez, Saab-Rincón, Reyes y Covarrubias 2016 ). Específicamente, el laboratorio de la Dra. Alejandra Covarrubias y el Dr. José Luis Reyes del Instituto de Biotecnología de la UNAM se ha enfocado en entender la función de las proteínas «hidrofilinas» durante la respuesta adaptativa de las plantas al déficit hídrico, así como el papel de estas proteínas en otros organismos eucariontes y procariontes. Asimismo, se están estudiando aspectos relativos a la participación de los microARN como reguladores de la respuesta genética a déficit hídrico, particularmente en frijol. Finalmente, se estudia cómo se da la regulación del metabolismo y la distribución de sacarosa durante la respuesta a sequía en plantas de frijol y cuál es el impacto en la productividad de este cultivo.
Los grupos de la Dra. Gladys-Cassab y del Dr. Jorge Nieto del Instituto de Biotecnología y el Jardín Botánico del Instituto de Biología, respectivamente, de la UNAM, están investigando la relación entre el manejo y la selección que realizan campesinos en la generación de variedades nativas de maíz resistentes a la sequía, con capacidad de germinar y emerger en condiciones de siembra profunda. Una de estas variedades es el llamado «maíz de cajete», cuya semilla puede ser sembrada a más de 30 cm de profundidad. A esta profundidad la planta germinada puede alcanzar la humedad residual del suelo y dado que desarrolla un mesocotilo particularmente largo, la plúmula puede emerger del sustrato exitosamente ( Nieto-Sotelo et al., 2016 ). Además, se están investigando las características fisiológicas y genéticas de estas variedades con fines de introgresar estas características a líneas mejoradas de mayor rendimiento ( Nieto-Sotelo et al., 2016).
Otras investigaciones relevantes que estudian cambios de desarrollo en plantas que crecen en ambientes áridos son las del laboratorio del Dr. Joseph Dubrovsky ( Dubrovsky y Gómez-Lomelí, 2003; Shishkova et al., 2013; Shishkova, Rost y Dubrovsky, 2008 ).
Variación natural de ecotipos de Arabidopsis : herramienta para estudios Eco-Evo-Devo
Los estudios con ecotipos de Arabidopsis han permitido explorar la gran variabilidad fenotípica que existe en diferentes poblaciones de esta planta, tanto en su morfología como en su desarrollo. Recientemente, el grupo de la Dra. María Paz Sánchez del Instituto de Ecología de la UNAM estudió la arquitectura de la raíz de 12 ecotipos de Arabidopsis provenientes de diferentes sitios geográficos con condiciones ambientales contrastantes, documentando una gran variabilidad en el desarrollo de este órgano entre ecotipos. Se encontró que existe una correlación entre la morfología de la raíz y las condiciones de humedad de los sitios de origen de algunos ecotipos. Tal resultado indica la existencia de una base molecular consistente que ha permitido la conservación de este carácter, aun en condiciones homogéneas de crecimiento en laboratorio ( Aceves-García et al., 2016). Por otra parte, Rosas et al. (2013) encontraron loci específicos asociados con la variación en la arquitectura de la raíz en ecotipos de Arabidopsis . Los avances en el conocimiento de las bases genéticas y epigenéticas de esta variación y el estudio de la interrelación con factores ambientales asociados serán una contribución importante para el desarrollo de la Eco-Evo-Devo en México.
Subdiferenciación celular en epidermis de Arabidopsis : un sistema modelo teórico-experimental para la Eco-Evo-Devo
En México se ha estudiado cómo una misma red de regulación genética puede estar involucrada en moldear patrones morfogenéticos contrastantes en órganos distintos de un mismo organismo ( Benítez et al., 2007, 2008; Mendoza y Álvarez-Buylla, 2000 ). A su vez, estos patrones se pueden alterar de manera plástica en respuesta a mutaciones o variaciones en las condiciones ambientales. Por ejemplo, la disposición y la densidad de tricoblastos (células encargadas de producir pelos de la raíz), se alteran plásticamente cuando se modifican, entre otros factores ambientales, la concentración de fosfatos, la cantidad de agua o de sales en el suelo ( Mendoza y Álvarez-Buylla, 2000 ). Por lo tanto, este módulo de desarrollo es un sistema que se ha usado para los estudios Eco-Evo-Devo ( Benítez, Azpeitia y Álvarez-Buylla, 2013 ).
En México también se ha estudiado cómo otras señales internas alteran el patrón de la morfología de los órganos, como ocurre con los pelos de la raíz de Arabidopsis (Benítez et al., 2013 ). Así, se ha analizado cómo el etileno puede actuar sobre la red de regulación genética implicada en la emergencia o no de pelo en la raíz ( Mendoza y Álvarez-Buylla, 2000 ) y cómo la vía de las giberelinas se retroalimenta de una vía de transducción de señales hormonales, lo cual podría estar actuando sobre el módulo de subdiferenciación epidérmica ( Benítez et al., 2008 ). También se ha encontrado que, al igual que en la raíz, el patrón de tricomas (pelos) en la epidermis de las hojas se modifica con tratamientos de fitohormonas ( Benítez et al., 2013 ) y por diversos factores ambientales.
Al contar con modelos de redes o módulos que subyacen al proceso de diferenciación celular y formación de patrones, como el que ocurre en la subdiferenciación epidérmica en Arabidopsis , es posible realizar estudios mecanísticos y sistémicos de Eco-Evo-Devo. Por ejemplo, se ha mostrado que genes implicados en el módulo que regula la formación de pelos epidérmicos también regulan la formación de la cutícula. Por lo tanto, mutantes que pierden la función de estos genes son más susceptibles, por ejemplo, al ataque de herbívoros. De manera interesante, se ha demostrado que esta función es recuperada parcialmente por la acción de giberelinas ( Xia et al., 2012 ). Este ejemplo muestra la importancia que tienen las vías de transducción de hormonas para las interacciones de los mecanismos que subyacen a la diferenciación celular, a la morfogénesis y a distintos fenómenos ecológicos.
El sistema regulatorio de subdiferenciación epidérmica también resulta instrumental para explorar las bases mecanísticas y sistémicas del origen de distintos fenotipos y de su variación. De manera complementaria, en un laboratorio del Departamento de Producción Agrícola y Animal de la Universidad Autónoma Metropolitana unidad Xochimilco, se estudia si es posible establecer una relación entre la variación fenotípica y la variación molecular de genes que intervienen en la red regulatoria implicada en la formación de pelos radiculares, y cómo esta última variación se correlaciona con los distintos patrones epidérmicos presentes en distintos ecotipos y linajes. Por ejemplo, se está indagando si las secuencias de los genes de la red de subdiferenciación epidérmica han evolucionado por selección natural y qué tan restringidas están por la red genética regulatoria en la que éstos participan (e.g., Dávila-Velderrain et al., 2014 ).
Hasta ahora, se cuenta con información preliminar acerca de los patrones epidérmicos en distintos ecotipos ( Telfer, Bollman y Poethig, 1997 ), sugiriendo que estos podrían ser adaptativos; aunque también pueden haberse fijado por presión sobre otros módulos con los cuales están acoplados. Estos patrones de variación fenotípica pueden alterar la ecología de las plantas, y esta variación también puede heredarse mediante mecanismos tanto genéticos como epigenéticos, impactando en la evolución de estos rasgos y en las poblaciones o especies que los portan. Este sistema también resulta muy útil para estudios de Eco-Evo-Devo porque parece que las alteraciones en los patrones de pelos y tricomas tienen claros impactos en la ecología de las plantas ( Benítez et al., 2013).
Un ejemplo interesante, que ilustra cómo se puede ir del estudio de genes particulares a las redes genéticas subyacentes y de aquí a los mecanismos de desarrollo, tiene que ver con las implicaciones ecológicas y evolutivas del patrón de tricomas y la resistencia al ataque de herbívoros ( Kivimäki, Kärkkäinen, Gaudeul, Løe y Agren, 2007 ). En 2 especies se ha descrito que las alteraciones epidérmicas causadas por herbivoría pueden heredarse. Este sistema podría usarse para explorar la asimilación genética y los mecanismos sistémicos involucrados ( Benítez et al., 2013 y referencias ahí citadas). La asimilación genética implica que modificaciones del desarrollo elicitadas por el ambiente pueden heredarse y eventualmente, impactar en la evolución de las especies. Esto podría proveer la validación experimental de propuestas teóricas que sugieren que la plasticidad del desarrollo es fundamental para la evolución adaptativa.
Evolución del desarrollo y el papel de la ecología en sistemas animales
En México existen especies animales que son consideradas organismos modelo para estudios de Evo-Devo y Eco-Evo-Devo. Una de estas especies es el pez ciego de ríos subterráneos, Astyanax mexicanus , el cual ha sido útil en el estudio de adaptaciones a la oscuridad. Debido a que existen especies cercanamente emparentadas a A. mexicanus que viven en cuerpos de agua superficiales y poseen ojos funcionales, se han desarrollado estudios comparativos dirigidos a entender las causas genéticas, epigenéticas y ambientales que subyacen a esta variación interespecífica en la función visual ( Santacruz et al., 2015). En la intersección de la epigenética, la variación críptica existente y la ecología, los estudios comparativos proponen que la variación críptica (es decir, variación genética que no se expresa) por la acción de un gen (chaperona HSP90) que, bajo condiciones ambientales nuevas (como someter a un régimen de oscuridad a especies de Astyanax que viven en cuerpos de agua superficiales), desestabiliza su acción homogenizadora exhibiendo la variación críptica presente en la especie y posibilitando la selección de peces con ojos progresivamente más pequeños ( Rohner et al., 2013).
También existen otras especies animales, tales como los organismos troglobios de diferentes cuerpos de agua subterráneos (como ríos y cenotes) y organismos marinos, que son de interés de estudios para la Eco-Devo-Evo y que están en desarrollo en México, tales como los que se llevan a cabo en el Instituto de Ciencias del Mar y Limnología de la UNAM. Un estudio sobresaliente es el de Caballero et al. (2012) en el que se propone un modelo que incorpora aspectos epigenéticos, de migración celular, de interacciones tisulares y de fuerzas mecánico-elásticas para explicar la variación de patrones de coloración de distintos linajes de peces, anfibios y reptiles. Otra área de estudio, llevada a cabo en el Instituto de Fisiología de la UNAM, se enfoca en entender las bases moleculares de los procesos cancerígenos en mamíferos. En particular, se estudian los mecanismos epigenéticos involucrados en la regulación de genes supresores de tumores, tales como aquellos relacionados con la regulación epigenética de microARN involucrados en el desarrollo de glioblastoma multiforme, un tipo de cáncer de las células gliales ( Ayala-Ortega et al., 2016).
Implicaciones de la Eco-Evo-Devo en la biomedicina
Una perspectiva metodológica de la Eco-Evo-Devo permite integrar un enfoque preventivo en la comprensión y el tratamiento de enfermedades crónico-degenerativas, tales como la diabetes tipo 2 (primera causa de muerte en México) y el cáncer en sus diversas manifestaciones. Estas enfermedades resultan de desajustes severos de procesos de regulación genética del organismo. La comprensión de estos procesos, para su potencial tratamiento terapéutico, exige una perspectiva holística que tome en cuenta aspectos genéticos, epigenéticos, ambientales y socioculturales que modulan las dinámicas biomoleculares y sus acoplamientos con los campos físico-químicos vitales que sustentan la salud humana. Esta perspectiva es importante para entender el papel crítico que tiene el estilo de vida (alimentación, actividad física y exposición a tóxicos, a infecciones y al estrés, principalmente) en la emergencia y progresión de la enfermedad, entendida esta como un desajuste nocivo, de los procesos de desarrollo y fisiológicos del organismo. La importancia del ambiente (e.g., estilo de vida) en las probabilidades de emergencia y de progresión de enfermedades degenerativas está cada vez mejor documentada ( World Health Organization, 2014).
En el contexto anterior se están desarrollando varios estudios en el Laboratorio de Genética Molecular, Epigenética, Desarrollo y Evolución de Plantas del Instituto de Ecología (UNAM). Un primer estudio trata de descubrir los módulos regulatorios, principalmente transcripcionales, que subyacen a los patrones genéticos genéricos, descritos a nivel celular y tisular, relacionados con una enfermedad. También se estudia la dinámica de interacción entre estos módulos y los procesos metabólicos que, por ejemplo, causan la inflamación de tejidos. Esta última se da en respuesta a factores ambientales que activan vías de transducción de señales (e.g., calcio, hormonas, etc.) que están conectadas con los módulos regulatorios que subyacen al funcionamiento de células efectoras. Estas células, y los sistemas a los que pertenecen, son fundamentales para la interacción del organismo con su ambiente o estilo de vida.
En el contexto de los procesos que afectan el balance entre la diferenciación y la proliferación celular, un grupo del Instituto Politécnico Nacional estudia los procesos que subyacen a la carcinogénesis epitelial ( Méndez-López et al., 2016 ). Otros grupos de investigación en México estudian, con enfoques de biología de sistemas, la leucemia linfoblástica aguda infantil ( Enciso, Mayani, Mendoza y Pelayo, 2016 ), que es una causa importante de mortalidad y parece estar más asociada con factores ambientales que con predisposiciones genéticas simples. También se ha estudiado el sistema inmunológico con enfoques sistémicos ( Martínez-Sánchez, Mendoza, Villarreal y Álvarez-Buylla, 2015; Mendoza, 2013 ).
En México es importante desarrollar estudios centrados en la comprensión y tratamiento de enfermedades desde la perspectiva de la Eco-Evo-Devo integrando 3 aspectos importantes: la exposición de la población a agentes nocivos, las consecuencias concretas de esquemas específicos de alimentación así como de tratamiento médico y, en general, la dinámica espacio-temporal del estilo de vida. Los resultados exitosos alcanzados hasta ahora en otras partes del mundo, bajo enfoques de protección de la salud de corte preventivo ( Harris, 2006 ), son muy prometedores. Es importante reconocer que las enfermedades crónico-degenerativas resultan de desajustes en el desarrollo apropiado del organismo, las cuales deben entenderse en un contexto ecológico y evolutivo más que en explicaciones basadas en alteraciones genéticas simples. La caracterización funcional de las dinámicas ambientales (biológicas y socioculturales, básicamente), que repercuten en los procesos que subyacen a la plasticidad fenotípica del organismo, es una elección terapéutica obvia ( Espinosa-Soto et al., 2011b).
El desarrollo de la investigación en Eco-Evo-Devo, en el contexto de la comprensión y tratamiento de enfermedades complejas, está teniendo cobijo en México. Así, se desarrollan seminarios y cursos especializados en el Centro de Ciencias de la Complejidad (C3) de la UNAM, en torno a la biología de sistemas de las enfermedades complejas y su relación transversal con los programas de ecología, de ciencias ambientales y de salud. En estas actividades participan investigadores y estudiantes provenientes de diversos entornos académicos e institucionales, colaborando bajo pautas multidisciplinarias. Así, la perspectiva de Eco-Evo-Devo, con enfoques sistémicos, pretende contribuir al sector salud y socioambiental y privilegiar una medicina preventiva, fomentando prácticas individuales, colectivas y comunitarias saludables.
Implicaciones de la Ecología Evolutiva del Desarrollo en la agricultura
Un aspecto fundamental para el mejoramiento genético de las plantas de importancia agrícola es el uso de modelos que permitan entender los mecanismos sistémicos de la herencia y cómo el ambiente puede influir en cambios fenotípicos dirigidos. Este tema ha sido históricamente el objeto de los esfuerzos en el mejoramiento agronómico. En este contexto, la cuestión del papel que desempeña la plasticidad fenotípica en la evolución adaptativa es de particular interés. Para el fitomejoramiento, es conveniente enfocarse en los rasgos y variedades que tienen normas de reacción con una amplia variación fenotípica en respuesta a gradientes ambientales que son relevantes a un entorno agrícola determinado. La posibilidad de contar con modelos sistémicos de la regulación genética subyacente a los rasgos de interés, en particular de aquellos donde se involucran múltiples loci, permitirá tener escenarios predictivos de éxito en los programas de mejoramiento agrícola.
Esta propuesta, que implica un enfoque más integrativo que el mejoramiento genético tradicional (enfocado en generar líneas puras endogámicas), implica conocer y aprovechar la diversidad genética y fenológica existente en poblaciones de variedades nativas. En México este enfoque se denomina mejoramiento masal, el cual ha sido abordado en el Colegio de Postgraduados por años, enfocándose en el mejoramiento de maíces nativos (e.g., Martínez-Zambrano, Galán, González y Muñoz, 2000 ). Dentro de este mejoramiento, algunas experiencias que parten de la diversidad de variedades nativas en la agricultura tradicional han dado muy buenos resultados aumentando el rendimiento de los cultivos ( Macchi-Leite, Rincón-Sánchez, Ruiz-Torres y Castillo-González, 2010 ) o la resistencia a plagas y sequía ( Martínez-Zambrano et al., 2000 ). Resulta muy interesante que los mejores rendimientos en maíz se observan en condiciones subóptimas, cuando las plántulas de maíz experimentan condiciones contrastantes de humedad, que en muchos casos ejercen una presión de selección que favorece la expresión de la «ganancia genética» acumulada a través de varias generaciones de selección masal ( Martínez-Zambrano et al., 2000 ).
Estudios futuros sobre cultivos domesticados en México, como los usados en el sistema de policultivo tipo milpa, pueden servir de modelos de estudios de la Eco-Evo-Devo para indagar, por ejemplo, qué rasgos son o pueden ser más susceptibles de evolucionar por efectos del ambiente (incluyendo, por ejemplo, el manejo campesino) y el porqué de tal susceptibilidad. Estos estudios pueden fortalecerse con el uso de modelos de redes complejas dinámicas que integren los componentes genéticos y no genéticos involucrados en la emergencia de los fenotipos bajo análisis. Un grupo de investigación de la UAM Xochimilco está desarrollando una línea de investigación en esta dirección.
Conclusiones y perspectivas
Los estudios de Eco-Evo-Devo en México pueden ayudar al desarrollo de este campo a través de: 1) expandir la base teórica existente, emergida del trabajo seminal de Waddington (1957), 2) aumentar las plataformas de simulación computacionales, incorporando aproximaciones y datos novedosos en sistemas modelo, 3) ampliar los estudios a otras especies, más allá de los sistemas modelo clásicos (basados principalmente en Arabidopsis), 4) contribuir en la formulación y estudio de modelos teóricos que sean dinámicos, integrativos de distintos niveles de organización biológica (redes de regulación genética, células, tejidos, órganos, organismos) y que consideren la interacción de estos niveles con el ambiente y 5) implementar enfoques experimentales que validen los modelos y analicen las implicaciones relevantes para México.
De particular interés, para un país megadiverso como México, será evaluar las inferencias hechas a partir del estudio de sistemas modelo con otras especies que presenten peculiaridades de desarrollo y/o fenotipos «extremos». Esto puede permitir establecer qué módulos del desarrollo, elementos genéticos y/o epigenéticos pudiesen haberse modificado en linajes con historias evolutivas y ecológicas contrastantes. La gran cantidad de estudios de sistemática y morfología existente sobre de la diversidad de especies de México ofrece excelentes oportunidades para estudios de Eco-Evo-Devo. Por ejemplo, será interesante analizar si la gran diversidad de especies de angiospermas es producto de modificaciones al interior de un módulo de desarrollo particular ( Almeida et al., 2014 ), o bien si está más relacionados con cambios en las dinámicas de acoplamiento de diferentes módulos de desarrollo, o con vías de transducción de señales intracelulares inducidas por ambientes particulares.
En México, un enfoque de Eco-Evo-Devo puede desempeñar un papel crítico para repensar los programas de conservación y mejoramiento de la biodiversidad, en general, y la agrobiodiversidad, en particular. En este último punto, es urgente una evaluación más rigurosa y completa a corto, mediano y largo plazo de la liberación al ambiente de organismos genéticamente modificados (OGMs). Este enfoque también podría impactar de manera muy importante en la biomedicina y en los programas de salud pública. Algunas preguntas pendientes por abordar son: ¿Cómo son los patrones de variación temporal de los factores ambientales que generan alteraciones en el desarrollo y en las respuestas fenotípicas plásticas? ¿Qué tipo de patrones temporales de dichos factores son necesarios para que se hereden rasgos que emergen como consecuencia de la interacción de los mecanismos de desarrollo y el ambiente? es decir, ¿Bajo qué condiciones ambientales puede ocurrir la asimilación genética y cuáles son los mecanismos de herencia involucrados? Estas interrogantes implican un entendimiento de la herencia como un proceso emergente de la retroalimentación entre los mecanismos sistémicos del desarrollo y el ambiente a lo largo de generaciones. Esta conceptualización de la herencia contrasta con la visión que implica mecanismos meramente informacionales y de «lectura» o «ejecución» de programas genéticos. Entonces, se puede pensar que la heredabilidad de un rasgo moldeado por el efecto de algún factor ambiental depende tanto de los mecanismos intraorganísmicos que lo subyacen, como de la persistencia transgeneracional de la acción de ese factor. El estudio de este tipo de herencia implica exploraciones teórico-experimentales de Eco-Evo-Devo de sistemas bien estudiados a nivel del desarrollo y de las redes genéticas regulatorias involucradas.
Agradecimientos
Agradecemos el apoyo logístico y técnico de Diana Romo. El trabajo que sustenta este estudio ha recibido el apoyo de los siguientes donativos: Conacyt: 240180, 180380, 2015-01-687. UNAM-DGAPA-PAPIIT: IN205517, IN203814, IN211516, y NPTC-PRODEP SEP-UAM-PTC-597 UAM-X-CA-21. Nos disculpamos por la omisión de otros estudios relevantes a la Eco-Evo-Devo en México que no se incluyen en esta revisión por falta de espacio o porque no eran de nuestro conocimiento hasta el momento de la publicación del presente trabajo.
Referencias
Aceves-García, P., Alvarez-Buylla, E. R., Garay-Arroyo, A., García-Ponce, B., Muñoz, R. and Sanchez M. P. (2016). Root Architecture Diversity and Meristem Dynamics in Different Populations of Arabidopsis thaliana. Front Plant Sci 7, 858.
Aldana, M., Balleza, E., Kauffman, S. and Resendiz, O. (2007). Robustness and evolvability in genetic regulatory networks. Journal of theoretical biology, 245, 433-448.
Almeida, A. M. R., Yockteng, R., Schnable, J., Alvarez-Buylla, E. R., Freeling, M. and Specht, C. D. (2014). Co-option of the polarity gene network shapes filament morphology in angiosperms. Scientific Reports, 4, 6194.
Alonso-Blanco, C., Aarts, M. G., Bentsink, L., Keurentjes, J. J., Reymond, M., Vreugdenhil, D. and Koornneef, M. (2009). What has natural variation taught us about plant development, physiology, and adaptation? Plant Cell 21, 1877-1896.
Altieri, M. (2000). Ecological impacts of transgenic crops on agroecosystem health. Ecosystem Health, 6, 13-23.
Álvarez-Buylla, E., Benítez, Espinosa-Soto, C. (2011). Mutually reinforcing patterning mechanisms. Nature Reviews Molecular and Cell Biology. 12:265-273.
Álvarez-Buylla, E. R., Liljegren, S. J., Pelaz, S., Gold, S. E., Burgeff, C., Ditta, G. S., Vergara-Silva, F. and Yanofsky, M. F. (2000a). MADS-box gene evolution beyond flowers: Expression in pollen, endosperm, guard cells, roots and trichomes. Plant Journal, 24(4), 457–466.
Álvarez-Buylla, E. R., Pelaz, S., Liljegren, S. J., Gold, S. E., Burgeff, C., Ditta, G. S., Ribas de Pouplana, L., Martínez-Castilla, L. and Yanofsky, M. F. (2000b). An ancestral MADS-box gene duplication occurred before the divergence of plants and animals. PNAS, 97, 5328–5333.
Álvarez-Buylla, E., Benítez, M., Balleza, E., Chaos, A., Espinosa-Soto, C. and Padilla-Longoria, P. (2007a) Gene regulatory network models for plant development. Current Opinion in Plant Biology, 10, 83–91.
Álvarez-Buylla, E. R., Benítez, M. and Espinosa-Soto, C. (2007b). Phenotypic evolution is restrained by complex developmental processes. HFSP Journal, 1(2), 99–103.
Álvarez-Buylla, E. R., Chaos, A., Aldana, M., Benítez, M., Cortes-Poza, Y., Espinosa-Soto, C., Hartasánchez, D. A., Lotto, R. B., Malkin, D., Santos, G. J. and Padilla-Longoria, P. (2008). Floral morphogenesis: stochastic explorations of a gene network epigenetic landscape. PloS One, 3, e3626.
Álvarez-Buylla, E. R., Azpeitia, E., Barrio, R., Benítez, M., y Padilla-Longoria, P. (2010a). From ABC genes to regulatory networks, epigenetic landscapes and flower morphogenesis: Making biological sense of theoretical approaches. Seminars in Cell and Developmental Biology, 21, 108-117.
Álvarez-Buylla, E., Benítez, M., Corvera-Poiré, A., Chaos Cador, Á., de Folter, S., Gamboa de Buen, A., Garay-Arroyo, A., García-Ponce, B., Jaimes-Miranda, F., Pérez-Ruiz, R. V., Piñeyro-Nelson, A. and Sánchez-Corrales, Y. (2010b). Flower Development. The Arabidopsis Book, e0127, 1-57p.
Álvarez-Buylla, E. R., Ambrose, B. A., Flores-Sandoval, E., Englund, M., Garay-Arroyo, A., García-Ponce, B., de la Torre-Bárcena, E., Espinosa-Matías, S., Martínez, E., Piñeyro-Nelson, A., Engström, P. and Meyerowitz, M. (2010c). B-Function Expression in the Flower Center Underlies the Homeotic Phenotype of Lacandonia schismatica (Triuridaceae). The Plant Cell, 22: 3543-3559.
Álvarez-Buylla, E. R., Dávila-Velderrain, J., Martínez-García, J. C. (2016). Elena R. Systems Biology Approaches to Development beyond Bioinformatics: Nonlinear Mechanistic Models Using Plant Systems. BioScience. doi: 10.1093/biosci/biw027.
Álvarez-Buylla, E. R., Piñeyro-Nelson, A., Turrent-Fernández, A., Nieto-Sotelo, J., Wegier, A., Alavez, V., Milán, L., Traavik, T. y Quist, D. (2013). Insuficiencias, Riesgos y Peligros de la Ingeniería Genética en la Agricultura: Alternativas a la trayectoria tecnológica predominante. En “El maíz en peligro ante los transgénicos” (Unión de Científicos Comprometidos con la Sociedad. ed), UCCS, Mexico 111- 117.
Ambrose, B. A., Espinosa-Matías, S., Vázquez-Santana, S., Vergara-Silva, F., Martínez, E., Márquez-Guzmán, J. and Álvarez-Buylla, E. R. (2006). Comparative developmental series of the Mexican triurids support a euanthial interpretation for the unusual reproductive axes of Lacandonia schismatica (Triuridaceae). American Journal of Botany, 93, 15-35.
Andrés, F. and Coupland, G. (2012). The genetic basis of flowering responses to seasonal cues. Nature Genetics, 13, 627-639.
Antoniou, M., Robinson, C, Fagan, J. (2012). GMO Myths and Truths: An evidence-based examination of the claims made for the safety and efficacy of genetically modified crops, . In Food Freedom (Earth Open Source, ed).
Ayala-Ortega, E., Arzate-Mejia, R., Perez-Molina, R., Gonzalez-Buendia, E., Meier, K., Guerrero, G., and Recillas-Targa, F. (2016). Epigenetic silencing of miR-181c by DNA methylation in glioblastoma cell lines. BMC Cancer, 16, 226.
Azpeitia, E. and Alvarez-Buylla, E. R. (2012). A complex systems approach to Arabidopsis root stem-cell niche developmental mechanisms: From molecules, to networks, to morphogenesis. Plant Molecular Biology. http://doi.org/10.1007/s11103-012-9954-6.
Azpeitia, E., Benítez, M., Vega, I., Villarreal, C. and Alvarez-Buylla, E. R. (2010). Single-cell and coupled GRN models of cell patterning in the Arabidopsis thaliana root stem cell niche. BMC Systems Biology, 4, 134.
Azpeitia, E. y García, M. (2014). Multiscale Modeling of Dynamic Systems for Evolution. In: Frontiers in Ecology, Evolution and Complexity. Benítez, M., Miramontes, O., and Valiente, A. (eds). Copit Arxives EditoraC3.
Azpeitia, E., Weinstein, N., Benitez, M., Mendoza, L. and Alvarez-Buylla, E. R. (2013). Finding Missing Interactions of the Arabidopsis thaliana Root Stem Cell Niche Gene Regulatory Network. Front Plant Sci, 4, 110.
Balleza, E., Alvarez-Buylla, E. R., Chaos, A., Kauffman, S., Shmulevich, I. and Aldana, M. (2008). Critical dynamics in genetic regulatory networks: examples from four kingdoms. PLoS One, 3, e2456.
Barrio, R. A., Hernández-Machado, A., Varez, C., Romero-Arias, J. R., Alvarez-Buylla, E. R. (2010). Flower Development as an Interplay between Dynamical Physical Fields and Genetic Networks. PLoS ONE, 5, e13523.
Barrio, R. A., Romero-Arias, J. R., Noguez, M. A., Azpeitia, E., Ortiz-Gutiérrez, E., Hernández-Hernández, V., Cortes-Poza, Y. and Álvarez-Buylla, E. R. (2013). Cell patterns emerge from coupled chemical and physical fields with cell proliferation dynamics: the Arabidopsis thaliana root as a study system. PLoS Comput Biol, 9, e1003026.
Beckie, H. J., Hall, L. M., Simard, M. J., Leeson, J. Y., Willenborg, C. J. (2010). A framework for postrelease environmental monitoring of second-generation crops with novel traits. Crop Science, 50, 1587-1604.
Beemster, G. T. and Baskin, T. I. (1998). Analysis of cell division and elongation underlying the developmental acceleration of root growth in Arabidopsis thaliana. Plant Physiol, 116, 1515-1526.
Belhaj, K., Chaparro-García, A., Kamoun, S., Patron, N., Nekrasov, V. (2015) Editing plant genomes with CRISPR/Cas9. Curr. Opin. Biotech, 32, 76-84.
Benbrook, C. (2012). Impacts of genetically engineered crops on pesticide use in the U.S. the first sixteen years. Environmental Sciences Europe 24, 99164-96242.
Benítez, M. and Alvarez-Buylla, E. R. (2010). Dynamic-module redundancy confers robustness to the gene regulatory network involved in hair patterning of Arabidopsis epidermis. BioSystems, 102, 11-15.
Benítez, M., Azpeitia, E. and Alvarez-Buylla, E. R. (2013). Dynamic models of epidermal patterning as an approach to plant eco-evo-devo. Current Opinion in Plant Biology, 16, 11-18.
Benítez, M., Espinosa-Soto, C., Padilla-Longoria, P. and Alvarez-Buylla, E. R. (2008). Interlinked nonlinear subnetworks underlie the formation of robust cellular patterns in Arabidopsis epidermis: a dynamic spatial model. BMC Systems Biology, 2, 98.
Benítez, M., Espinosa-Soto, C., Padilla-Longoria, P., Díaz, J. and Alvarez-Buylla, E. R. (2007). Equivalent genetic regulatory networks in different contexts recover contrasting spatial cell patterns that resemble those in Arabidopsis root and leaf epidermis: A dynamic model. International Journal of Developmental Biology, 51, 139-155.
Benyshek, D. C. (2013). The “early life” origins of obesity-related health disorders: New discoveries regarding the intergenerational transmission of developmentally programmed traits in the global cardiometabolic health crisis. American Journal of Physical Anthropology, 152, 79-93.
Berger, S. L., Kouzarides, T., Shiekhattar, R., and Shilatifard, A. (2009). An operational definition of epigenetics. Genes Dev, 23, 781-783.
Berry, S. and Dean, C. (2015). Environmental perception and epigenetic memory: mechanistic insight through FLC. Plant J, 83, 133-148.
Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K. and Scheres, B. (2005). The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature, 433, 39-44.
Bortesi, L. and Fischer, R. (2015). The CRISPR/Cas9 system for plant genome editing and beyond. Biotechnology Advances 33, 41-52.
Burgeff, C., Huerta, E., Acevedo, F., Sarukhán, J. (2014) How much can GMO and non-GMO cultivars coexist in a megadiverse country? AgBioForum 17, 90-101.
Burssens, S., Himanen, K., van de Cotte, B., Beeckman, T., Van Montagu, M., Inzé, D. and Verbruggen, N. (2000). Expression of cell cycle regulatory genes and morphological alterations in response to salt stress in Arabidopsis thaliana. Planta, 211, 632-40.
Caballero, L., Benítez, M., Alvarez-buylla, E. R., Hernández, S., Arzola, A. V., y Cocho, G. (2012). An Epigenetic Model for Pigment Patterning Based on Mechanical and Cellular Interactions. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution, 318, 209-223.
Cecchini, E., Al-Kaff, N., Bannister, A., Giannakou, M., McCallum, D., Maule, A., Milner, J., Covey, S. (1998). Pathogenic interactions between variants of cauliflower mosaic virus and Arabidopsis thaliana. J Exp Bot, 49, 731-737.
Chaos, A., Aldana, M., Espinosa-Soto, C., de Leon, B. G. P., Arroyo, A. G. and Alvarez-Buylla, E. R. (2006). From genes to flower patterns and evolution: Dynamic models of gene regulatory networks. Journal of Plant Growth Regulation, 25, 278-289.
Chazen, O. and Neumann, P. M. (1994). Hydraulic signals from the roots and rapid cell-wall hardening in growing maize (Zea mays L.) leaves are primary responses to polyethylene glycol-induced water deficits. Plant Physiol, 104, 1385–1392.
Chen, H., Bodulovic, G., hall, P., Moore, A., Higgins, T., Djordjevic, M. and Rolfe, B. (2009). Unintended changes in protein expression revealed by proteomic analysis of seeds from transgenic pea expressing a bean a-amylase inhibitor gene. Proteomics 9, 4406-4415.
Clark, N. M., de Luis Balaguer, M. A. and Sozzani, R. (2014). Experimental data and computational modeling link auxin gradient and development in the Arabidopsis root. Front Plant Sci, 7, 328.
Comstock, J. P. (2002). Hydraulic and chemical signalling in the control of stomatal conductance and transpiration. J Exp Bot, 53, 195-200.
Couot-Gastelier, J. and Vartanian, N. (1995). Drought-induced Short Roots in Arabidopsis thaliana: Structural Characteristics. Botanica Acta, 108, 407–413.
Davies, W. J. and Zhang, J. H. (1991). Root signals and the regulation of growth and development of plants in drying soil. Ann Rev of Plant Physiol and Plant Molec Biol, 42, 55–76.
Davila-Velderrain, J. and Alvarez-Buylla, E. R. (2014). Bridging genotype and phenotype. Frontiers in Ecology, Evolution and Complexity, CopIt ArXives, 144-154.
Davila-Velderrain, J., Juarez-Ramiro, L., Martinez-Garcia, J.C. and Alvarez-Buylla, E.R. (2015a). Methods for Characterizing the Epigenetic Attractors Landscape Associated with Boolean Gene Regulatory Networks. arXiv preprint arXiv:1510.04230.
Davila-Velderrain, J., Martinez-Garcia, J. C. and Alvarez-Buylla, E. R. (2014a). Epigenetic Landscape Models: The Post-Genomic Era. bioRxiv, 4192.
Davila-Velderrain, J., Martinez-Garcia, J. C. and Alvarez-Buylla, E. R. (2015b). Modeling the epigenetic attractors landscape: toward a post-genomic mechanistic understanding of development. Frontiers in Genetics, 6, 160.
Davila-Velderrain, J., Martinez-Garcia, J. C. and Alvarez-Buylla, E. R. (2015c). Descriptive vs. Mechanistic Network Models in Plant Development in the Post-Genomic Era. Methods Mol Biol, 1284, 455-79.
Davila-Velderrain, J., Martinez-Garcia, J. C. and Alvarez-Buylla, E. R. (2016). Dynamic network modelling to understand flowering transition and floral patterning. Journal of Experimental Botany, 67, 2565-2572.
Davila-Velderrain, J., Servin-Marquez, A. and Alvarez-Buylla, E. R. (2014b). Molecular evolution constraints in the floral organ specification gene regulatory network module across 18 angiosperm genomes. Molecular Biology and Evolution, 31, 560-73.
Davila-Velderrain, J., Villarreal, C. and Alvarez-Buylla, E. R. (2015). Reshaping the epigenetic landscape during early flower development: induction of attractor transitions by relative differences in gene decay rates. BMC Systems Biology, 9, 1.
De Marchis, F., Valeri, M., Pompa, A., Bouveret, E., Alagna, F., Grisan, S., Stanzione, V., Mariotti, R., Cultrera, N., Baldoni, L. and Belluci, M. (2016). Overexpression of the olive acyl carrier protein gene (OeACP1) produces alterations in fatty acid composition of tobacco leaves. Transgenic Research, 25, 45-61.
Díaz, J. and Alvarez-Buylla, E. R. (2006). A model of the ethylene signaling pathway and its gene response in Arabidopsis thaliana: pathway cross-talk and noise-filtering properties. Chaos, 16, 23112.
Díaz, J. and Alvarez-Buylla, E. R. (2009). Information flow during gene activation by signaling molecules: ethylene transduction in Arabidopsis cells as a study system. BMC Systems Biology, 3, 48.
Dinneny, J. R., Long, T. A., Wang, J. Y., Jung, J. W., Mace, D., Pointer, S., Barron, C., Brady, S. M., Schiefelbein, J. and Benfey, P. N. (2008). Cell identity mediates the response of Arabidopsis roots to abiotic stress. Science, 320, 942-945.
Dobzhansky, T. (1937). Genetics and the Origin of Species. Columbia University Press.
Dyer, G. A., Serratos-Hernandez, J. A., Perales, H. R., Gepts, P., Pineyro-Nelson, A., Chavez, A., Salinas-Arreortua, N., Yunez-Naude, A., Taylor, J. E., and Alvarez-Buylla, E. R. (2009). Dispersal of transgenes through maize seed systems in Mexico. PloS One, 4, e5734.
Egger, G., Liang, G., Aparicio, A., and Jones, P.A. (2004). Epigenetics in human disease and prospects for epigenetic therapy. Nature, 429, 457-463.
Ellstrand, N. (2003). Current knowledge of gene flow in plants: implications for transgene flow. Phli Trans R Soc Lond, 358, 1163-1170.
Enciso, J., Mayani, H., Mendoza, L. and Pelayo, R. (2016). Modeling the Pro-inflammatory Tumor Microenvironment in Acute Lymphoblastic Leukemia Predicts a Breakdown of Hematopoietic-Mesenchymal Communication Networks. Frontiers in Physiology, 7, 349.
Espinosa-Matías, S.; Vergara-Silva, F.; Vázquez-Santana, S.; Martínez-Zurita, E.; Márquez Guzmán, J. 2012. Complex patterns of morphogenesis, embryology, and reproduction in Triuris brevistylis, a species of Triuridaceae (Pandanales) closely related to Lacandonia schismatica. Botany., 90: 1133-1151.
Espinosa‐Soto, C. (2016). Selection for distinct gene expression properties favours the evolution of mutational robustness in gene regulatory networks. Journal of Evolutionary Biology. (en prensa).
Espinosa-Soto, C., Immink, R. G., Angenent, G. C., Alvarez-Buylla, E. R. and de Folter, S. (2014). Tetramer formation in Arabidopsis MADS domain proteins: analysis of a protein-protein interaction network. BMC Syst Biol, 8, 9.
Espinosa‐Soto, C., Martin O. C., Wagner, A. (2011a). Phenotypic robustness can increase phenotypic variability after nongenetic perturbations in gene regulatory circuits. Journal of Evolutionary Biology, 24, 1284-97.
Espinosa-Soto, C., Martin, O. C, and Wagner, A. (2011b). Phenotypic plasticity can facilitate adaptive evolution in gene regulatory circuits. BMC evolutionary biology, 11, 5.
Espinosa-Soto, C., Martin, O. C., and Wagner, A. (2011c). Phenotypic plasticity can facilitate adaptive evolution in gene regulatory circuits. BMC Evolutionary Biology, 11, 5.
Espinosa-Soto, C., Padilla-Longoria, P. and Alvarez-Buylla, E. R. (2004). A gene regulatory network model for cell-fate determination during Arabidopsis thaliana flower development that is robust and recovers experimental gene expression profiles. Plant Cell, 16, 2923–2939.
Espinosa-Soto C, Wagner A. (2010). Specialization can drive the evolution of modularity. PLoS Comput Biol., 6, e1000719.
Feng, S., Jacobsen, S. E. and Reik, W. (2010). Epigenetic reprogramming in plant and animal development. Science 330, 622-627.
Fisher, R. (1930). The Genetical Theory of Natural Selection. Oxford University Press, 292 pp.
Frensch, J. and Hsiao, T. C. (1994). Transient Responses of Cell Turgor and Growth of Maize Roots as Affected by Changes in Water Potential. Plant Physiol, 104, 247-254.
Friml, J., Vieten, A., Sauer, M., Weijers, D., Schwarz, H., Hamann, T., Offringa, R. and Jürgens, G. (2003). Efflux-dependent auxin gradients establish the apical-basal axis of Arabidopsis. Nature, 426, 147-53.
Galinha, C., Hofhuis, H., Luijten, M., Willemsen, V., Blilou, I., Heidstra, R. and Scheres, B. (2007). PLETHORA proteins as dose-dependent master regulators of Arabidopsis root development. Nature, 449, 1053-7.
Galinha, C., Bilsborough, G. and Tsiantis, M. (2009). Hormonal input in plant meristems: A balancing act. Semin Cell Dev Biol, 20, 1149-56.
Garay-Arroyo, A., Sánchez, M. P., García-Ponce, B., Azpeitia, E. and Alvarez-Buylla, E.R. (2012). Hormone symphony during root growth and development. Dev Dyn, 241, 1867-85.
Garay-Arroyo, A., Ortiz-Moreno, E., de la Paz Sanchez, M., Murphy, A. S., Garcia-Ponce, B., Marsch-Martinez, N., de Folter, S., Corvera-Poiré, A., Jaimes-Miranda, F., Pacheco-Escobedo, M. A., Dubrovsky, J. G., Pelaz, S. and Alvarez-Buylla, E. R. (2013). The MADS transcription factor XAL2/AGL14 modulates auxin transport during Arabidopsis root development by regulating PIN expression. The EMBO Journal, 32, 2884-95.
García-Cruz, K. V., García-Ponce, B., Garay-Arroyo, A., Sanchez, M. P., Ugartechea-Chirino, Y., Desvoyes, B., Pacheco-Escobedo M. A., Tapia-López, R., Ransom-Rodríguez, I., Gutierrez, C. and Alvarez-Buylla, E. R. (2016). The MADS-box XAANTAL1 increases proliferation at the Arabidopsis root stem-cell niche and participates in transition to differentiation by regulating cell-cycle components. Annals of Botany, mcw126.
Gilbert, S. F. (2012). Ecological developmental biology: environmental signals for normal animal development. Evolution and development, 14, 20-28.
Gilbert, S. F. and Epel, D. (2009). Ecological Developmental biology: Integrating Epigenetics, Medicine and Evolution. Sinauer Associated, Inc.
Goldbergh, K. B., Kiernan, J. and Sheperd, R. (1991). A disease syndrome associated with expression of gene VI of Caulimoviruses may be a nonhost reaction. Molec Plant -Microbe Interactions 4, 182-189.
Goodwin, B. (1994). How the Leopard Changed its Spots: The Evolution of Complexity, Scribner, Princeton University Press, 252 pp.
Haldane, J. B. S. (1932). The Causes of Evolution. Longman, Green and Co. reimpresión de la Princeton University Press.
Hales, C. N. and Barker, D. J. P. (2013). Type 2 (non-insulin-dependent) diabetes mellitus: the thrifty phenotype hypothesis. International Journal of Epidemiology, 42, 1215-1222.
Han, P., García-Ponce, B., Fonseca-Salazar, G., Alvarez-Buylla, E. R. and Yu, H. (2008). AGAMOUS-LIKE 17, a novel flowering promoter, acts in a FT independent photoperiod pathway. Plant J., 55, 253-265.
Hapiak, M., Li, Y., Agama, K., Swade, S., Okenka, G., Falk, J., Khandekar, S., Raikhy, G., Anderson, A., Pollock, J., Zellner, W., Schoelz, J. and Leisner, S. (2008). Cauliflower mosaic virus gene VI product N-terminus contains regions involved in resistance-breakage, self-association and interactions with movement protein. Virus Res, 138, 119-129.
Harris, J. (2006). Keeping Cuba healthy. BBC News.
Heard, E. and Martienssen, R. A. (2014). Transgenerational epigenetic inheritance: Myths and mechanisms. Cell, 157, 95-109.
Hernández-Hernández, T., Martínez-Castilla, L. P. and Alvarez-Buylla, E. R. (2007). Functional diversification of B MADS-box homeotic regulators of flower development: Adaptive evolution in protein-protein interaction domains after major gene duplication events. Molecular Biology and Evolution, 24, 465–481.
Hernández-Hernández, V., Rueda, D., Caballero, L., Alvarez-Buylla, E. R. and Benítez, M. (2014). Mechanical forces as information: an integrated approach to plant and animal development. Front Plant Sci, 10, 265.
Hsiao, T. C. and Xu, L. K. (2000). Sensitivity of growth of roots versus leaves to water stress: biophysical analysis and relation to water transport. J Exp Bot, 51, 1595-616.
Ho, M. (1998). Fatal flaws in food safety assessment: critique of the joint FAO/WHO biotechnology and food safety report. Environmental and Nutritional Interactions, 2, 51-84.
Hsu, P., Lander, E., Zhang, F. (2014) Development and applications of CRISPR-Cas9 for genome engineering. Cell, 157, 1262-1278.
Jablonka, E., and Lamb, M. J. (2002). The changing concept of epigenetics. Ann N Y Acad Sci. 981, 82-96.
Jablonka, E. and Lamb, M. J. (2005). Evolution in four dimensions: Genetic, epigenetic, behavioral and symbolic variation in the history of life. Cambridge, Massachusetts, USA. MA: MIT press.
Jablonka, E. and Raz, G. (2009). Transgenerational epigenetic inheritance: prevalence, mechanisms, and implications for the study of heredity and evolution. The Quarterly review of biology, 84, 131-176.
James, C. (2014). Global Status of Commercialized Biotech/GM Crops: 2014. In ISAAA Brief, Ithaca, NY.
Johal, G. S. and Huber, D. M. (2009). Glyphosate effects on diseases of plants. Eur Journal of Agronomy, 31, 144-152.
Johanson, U., West, J., Lister, C., Michaels, S. D., Amasino, R. M. and Dean, C. (2000). Molecular analysis of FRIGIDA, a major determinant of natural varition in Aradopsis flowering time. Science, 290, 344-347.
Jones, A. L. and Sung, S. (2014). Mechanisms underlying epigenetic regulation in Arabidopsis thaliana. Integr Comp Biol, 54, 61-67.
Karlsson, B. H., Sills, G. R. and Nienhuis, J. (1993). Effects of photoperiod and vernalization on the number of leaves at flowering in 32 Arabidopsis thaliana (Brassicaceae) ecotypes. Am. J. Bot., 80, 646–48.
Kauffman, Stuart (1969). “Metabolic stability and epigenesis in randomly constructed genetic nets”. Journal of Theoretical Biology. 22 (3): 437–467. doi:10.1016/0022-5193(69)90015-0. PMID 5803332.
Kivimäki, M., Kärkkäinen, K., Gaudeul, M., Løe, G. and Agren, J. (2007). Gene, phenotype and function: GLABROUS1 and resistance to herbivory in natural populations of Arabidopsis lyrata. Mol Ecol, 16, 453-462.
Koornneef, M., Alonso-Blanco, C. and Vreugdenhil, D. (2004). Naturally occurring genetic variation in Arabidopsis thaliana. Annu Rev. Plant Biol., 55, 141-172.
Kouzarides, T. (2007). Chromatin modifications and their function. Cell 128, 693-705.
Lawton-Rauh, A., Alvarez-Buylla, E. and Purugganan, M. (2000). Molecular evolution of flower development. Trends in Ecology y Evolution (Personal Edition), 15, 144-149.
Lewontin, R. C. (1974). The genetic basis of evolutionary change. New York: Columbia University Press.
Lövei G., A., D.A., Arpaia, S. (2009). Transgenic insecticidal crops and natural enemies: a detailed review of laboratory studies. Envirenmental Entomology, 38, 293-306.
Liu, Y., Geyer, R., Van Zanten, M., Carles, A., Li, Y., Horold, A., Van Nocker, S. and Soppe, W. J. (2011). Identification of the Arabidopsis REDUCED DORMANCY 2 gene uncovers a role for the polymerase associated factor 1 complex in seed dormancy. PLoS One 6, e22241.
Macchi Leite, G., Rincón Sánchez, F., Ruiz Torres, N. A. and Castillo González, F. (2010). Selección y mantenimiento de poblaciones. Una perspectiva para la conservación in situ de la diversidad genética del maíz. Revista fitotecnia mexicana, 33(spe4), 43-47.
Mähönen, A. P., ten Tusscher, K., Siligato, R., Smetana, O., Díaz-Triviño, S., Salojärvi, J., Wachsman, G., Prasad, K., Heidstra, R. and Scheres, B. (2014). PLETHORA gradient formation mechanism separates auxin responses. Nature, 515, 125-9.
Marcellini, S., González, F., Sarrazin, A. F., Pabón Mora, N., Benítez, M., Piñeyro-Nelson, A. and Da Fonseca, R. N. (2016). Evolutionary Developmental Biology (EvoDevo) Research in Latin America. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution. (en prensa).
Martinez-Castilla, L. P. and Alvarez-Buylla, E. R. (2003). Adaptive evolution in the Arabidopsis MADS-box gene family inferred from its complete resolved phylogeny. PNAS, 100, 13407-12.
Martinez-Sanchez, M. E., Mendoza, L., Villarreal, C. and Alvarez-Buylla, E. R. (2015). A Minimal Regulatory Network of Extrinsic and Intrinsic Factors Recovers Observed Patterns of CD4+ T Cell Differentiation and Plasticity. PLOS Computational Biology, 11, e1004324.
Martínez-Zambrano, G. M., Galán, J. D. M., González, F. C. and Muñoz, M. L. (2000). Magnitud y linealidad de la respuesta a la selección masal en Maíz en función de los ambientes de selección y de evaluación. Agrociencia, 34, 429-436.
McCree, K. J. and Davis, S. D. (1974). Effect of Water Stress and Temperature on Leaf Size and on Size and Number of Epidermal Cells in Grain Sorghum. Crop Sci, 14, 751-755.
Méndez-López, L. F., Davila-Velderrain, J., Enríquez-Olguín, C., Domínguez-Hüttinger, E., Martinez-Garcia, J. C. and Alvarez-Buylla, E. R. (2016). Dynamic network and epigenetic landscape model of a regulatory core underlying spontaneous immortalization and epithelial carcinogenesis, 11. Molecular Networks. Retrieved from http://arxiv.org/abs/1604.02208
Mendoza, L. (2013). A Virtual Culture of CD4+ T Lymphocytes. Bulletin of Mathematical Biology, 75, 1012–1029.
Mendoza, L. and Alvarez-Buylla, E. R. (1998). Dynamics of the genetic regulatory network for Arabidopsis thaliana flower morphogenesis. J Theor Biol, 193, 307-319.
Mendoza, L. and Alvarez-Buylla, E. R. (2000). Genetic regulation of root hair development in Arabidopsis thaliana: A network model. Journal of Theoretical Biology, 204, 311–326.
Mendoza, L., Thieffry, D. and Alvarez-Buylla, E. R. (1999). Genetic control of flower morphogenesis in Arabidopsis thaliana: a logical analysis. Bioinformatics (Oxford, England), 15, 593-606.
Michaels, S. D. and Amasino, R. M. (1999). FLOWERING LOCUS C encodes a novel MADS domain protein that acts as a repressor of flowering. Plant Cell 11, 949-956.
Molitor, A. M., Bu, Z., Yu, Y., and Shen, W. H. (2014). Arabidopsis AL PHD-PRC1 complexes promote seed germination through H3K4me3-to-H3K27me3 chromatin state switch in repression of seed developmental genes. PLoS Genet 10, e1004091.
Motta S. S., Cluzel, P. and Aldana, M. (2015). Adaptive resistance in bacteria requires epigenetic inheritance, genetic noise, and cost of efflux pumps. PloS One, 10, e0118464.
Nakamich, N. (2015). Adaptation to the Local Environment by Modifications of the Photoperiod Response in Crops. Plant Cell Physiol, 56, 594-604.
Neumann, P. M. (1993). Rapid and reversible modifications of extension capacity of cell walls in elongating maize leaf tissues responding to root addition and removal of NaCl. Plant Cell and Environment, 16, 1107-1114.
Nieto-Sotelo, J., Vázquez L., Villa J. M., Ávila A. X., Rojas C. I., Aguilar C., Rangel L. M., Pérez S., Babu, R., Trachsel S., Zhang X. and Cassab G. I. (2016). Importance of mesocotyl and plumule growth on heat and drought avoidance in modern maize hybrids: physiology and GWAS. En: 58th Annual Maize Genetics Conference (memorias) Florida, EE.UU. 17-20 marzo p. 61.
Pérez-Ruiz, R. V., García-Ponce, B., Marsch-Martínez, N., Ugartechea-Chirino, Y., Villajuana-Bonequi, M., de Folter, S., Azpeitia, E., Dávila-Velderrain, J., Cruz-Sánchez, D., Garay-Arroyo, A., Sánchez, M. P., Estévez-Palmas, J. M., and Alvarez-Buylla, E. R. (2015). XAANTAL2 (AGL14) is an important component of the complex gene regulatory network that underlies Arabidopsis shoot apical meristem transitions. Molecular Plant, 8, 796-813.
Perrot-Rechenmann, C. (2010). Cellular responses to auxin: division versus expansion. Cold Spring Harb. Perspect. Biol, 2, a001446.
Pigliucci, M. (2003). Phenotypic integration: studying the ecology and evolution of complex phenotypes. Ecology Letters, 6, 265-72.
Pigliucci, M., Murren, C. J. and Schlichting, C. D. (2006). Phenotypic plasticity and evolution by genetic assimilation. J. Ex.p Biol., 209, 2362-2367.
Piñeyro-Nelson, A., Van Heerwaarden, J., Perales, H. R., Serratos-Hernández, J. A. Rangel, A., Hufford, M. B., Gepts, P., Garay-Arroyo, A., Rivera-Bustamante, R. and Alvarez-Buylla, E.R. (2009). Resolution of the Mexican transgene detection controversy: error sources and scientific practice in commercial and ecological contexts. Molecular Ecology, 18, 4145-4150.
Podevin, N. and Jardin, P. (2012). Possible consequences of the overlap between the CAMV 35S promoter regions in plant transformation vectors used and the viral gene VI in transgenic plants. GM crops and Food: Biotechnology and the Food Chain, 3, 296-300.
Rao, J., Yang, L., Guo, J., Quan, J., Chen, G., Zhao, X., Zhang, D., Shi, J. (2016). Metabolic changes in transgenic maize mature seeds over-expressing the Aspergillus niger phyA2. Plant Cell Rep, 35, 429-437.
Reiß, C., Olsson, L. and Hoßfeld, U. (2015). The history of the oldest self‐sustaining laboratory animal: 150 years of axolotl research. Journal of Experimental Zoology Part B: Molecular and Developmental Evolution, 324, 393-404.
Richards, E. J. (2006). Inherited epigenetic variation-revisiting soft inheritance. Nat Rev Genet 7, 395-401.
Rodriguez-Mega, E., Piñeyro-Nelson, A., Gutierrez, C., Garcia-Ponce, B., Sanchez, M. D. L. P., Zluhan-Martinez, E., Álvarez-Buylla, E.R. and Garay-Arroyo, A. (2015). Role of transcriptional regulation in the evolution of plant phenotype: A dynamic systems approach. Developmental Dynamics, 244, 1074-1095.
Rohner, N., Jarosz, D. F., Kowalko, J. E., Yoshizawa, M., Jeffery, W. R., Borowsky, R. L., and Tabin, C. J. (2013). Cryptic variation in morphological evolution: HSP90 as a capacitor for loss of eyes in cavefish. Science, 342, 1372-1375.
Rosas, U., Cibrian-Jaramillo, A., Ristova, D., Banta, J. A., Gifford, M. L., Fan, A. H., Zhou, R. W., Kim, G. J., Krouk, G., Birnbaum, K. D., Purugganan, M. D. and Coruzzi, G. M. (2013). Integration of responses within and across Arabidopsis natural accessions uncovers loci controlling root systems architecture. Proc Natl Acad Sci U S A, 110, 15133-15138.
Rymen, B., Coppens, F., Dhondt, S., Fiorani, F. and Beemster, G. T. (2010). Kinematic analysis of cell division and expansion. Methods Mol Biol, 655, 203-227.
Salazar-Henao, J. E., Vélez-Bermúdez, I. C., Schmidt, W., (2016). The regulation and plasticity of root hair patterning and morphogenesis. Development, 143, 1848-58.
Sanchez, M. P., Aceves-Garcia, P., Petrone, E., Steckenborn, S., Vega-Leon, R., Alvarez-Buylla, E.R., Garay-Arroyo, A., and Garcia-Ponce, B. (2015). The impact of Polycomb group (PcG) and Trithorax group (TrxG) epigenetic factors in plant plasticity. New Phytol., 208, 684-694.
Santacruz A. G., Tinoco-Cuellar O. M., Rangel-Huerta M., Maldonado E. (2015). Spatial Mapping in Perpetual Darkness: EvoDevo of Behavior in Astyanax mexicanus Cavefish. In: Keene A. C., Yoshizawa M, Mc-Gaugh S. E. Editors. Biology and evolution of the Mexican cavefish. 1st Edition, United States Elsevier p. 361–376.
Santner, A. and Estelle, M. (2009). Recent advances and emerging trends in plant hormone signalling. Nature, 459, 1071-1078.
Santner, A., Calderon-Villalobos, L.I. and Estelle, M. (2009). Plant hormones are versatile chemical regulators of plant growth. Nat Chem Biol, 5, 301-307.
Schuettengruber, B., Martinez, A.M., Iovino, N., and Cavalli, G. (2011). Trithorax group proteins: switching genes on and keeping them active. Nat Rev Mol Cell Biol, 12, 799-814.
Serratos-Hernandez, J. (2009). Bioseguridad y dispersión de maíz transgénico en México. Revista Ciencias, 92-93, 130-141.
Serratos-Hernández, J. A., Gómez-Olivares, J. L., Salinas-Arreortua, N., Buendía-Rodríguez, E., Islas-Gutiérrez, F., de Ita, A. (2007). Transgenic proteins in maize in the soil conservation area of Federal District, México. Frontiers in Ecology and the Environment, 5, 247-252.
Serratos-Hernández, J.A., Castillo-González, F., Kato-Yamakake, T.Á., Gómez-Olivares, J.L., Morales Valderrama. C. (2016). Conservación de la diversidad y la cultura del maíz nativo en el suelo de conservación. En: La biodiversidad en la Ciudad de México, vol. ii conabio/sedema. México, pp. 545-556. En prensa.
Sharp, R. E. and Davies, W. J. (1979). Solute regulation and growth by roots and shoots of water-stressed maize plants. Planta, 147, 43-49.
Shindo, C., Aranzana, M. J., Lister, C., Baxter, C., Nicholls, C., Nordborg, M. and Dean, C. (2005). Role of FRIGIDA and FLOWERING LOCUS C in determining variation in flowering time of Arabidopsis. Plant Physiol, 138, 1163-1173.
Shindo, C., Lister, C., Crevillen, P., Nordborg, M. and Dean, C. (2006). Variation in the epigenetic silencing of FLC contributes to natural variation in Arabidopsis vernalization response. Genes Dev., 20, 3079-3083.
Skirycz, A., Claeys, H., De Bodt, S, Oikawa, A., Shinoda, S., Andriankaja, M., Maleux, K., Eloy, N. B., Coppens, F., Yoo, S. D., Saito, K. and Inzé, D. (2011). Pause-and-stop: the effects of osmotic stress on cell proliferation during early leaf development in Arabidopsis and a role for ethylene signaling in cell cycle arrest. Plant Cell, 23, 1876-88.
Song, J., Angel, A., Howard, M. and Dean, C. (2012). Vernalization – a cold-induced epigenetic switch. J Cell Sci, 125, 3723-3731.
Suneson, C. A. (1956). An evolutionary plant breeding method. Agronomy Journal, 48, 188-191.
Tapia-López, R., García-Ponce, B., Dubrovsky, J. G., Garay-Arroyo, A., Pérez-Ruíz, R. V., Kim, S. H., Acevedo, F., Pelaz, S. and Alvarez-Buylla, E. R. (2008). An AGAMOUS-related MADS-box gene, XAL1 (AGL12), regulates root meristem cell proliferation and flowering transition in Arabidopsis thaliana. Plant Physiol, 146, 1182-1192.
Telfer, A., Bollman, K. M. and Poethig, R. S. (1997). Phase change and the regulation of trichome distribution in Arabidopsis thaliana. Development, 124, 645-654.
Torres-Sosa, C., Huang, S. and Aldana, M. (2012). Criticality is an emergent property of genetic networks that exhibit evolvability. PLoS Comput Biol., 8, e1002669.
Van Haperen, G. B. and Jacobs, J. (2012). Reconstruction of the Ethical debate oon naturalness in discussions about plant biotechnology. J Agric Environ Ethics, 25, 797-812.
Vázquez‐Lobo, A.; Carlsbecker, A.; Vergara‐Silva, A.; Alvarez‐Buylla, R.E.; Piñero, D.; and Engström, P. (2007). Characterization of the expression patterns of LEAFY/FLORICAULA and NEEDLY orthologs in female and male cones of the conifer genera Picea, Podocarpus, and Taxus: implications for current evo‐devo hypotheses for gymnosperms. Evolution and Development. 9:446-459.
Veenendaal, M. V. E., Painter, R. C., De Rooij, S. R., Bossuyt, P. M. M., Van Der Post, J. A. M., Gluckman, P. D. and Roseboom, T. J. (2013). Transgenerational effects of prenatal exposure to the 1944-45 Dutch famine. BJOG: An International Journal of Obstetrics and Gynaecology, 120, 548–553.
Vergara-Silva, F. (2003). Plants and the conceptual articulation of evolutionary developmental biology. Biology and Philosophy. 18: 249-284.
Vergara-Silva, F.; Martinez-Castilla, L. and Alvarez-Buylla, E. R. (2000). MADS-box genes: Development and evolution of plant body plans. Journal of Phycology. 36 (5), 803-812.
Vergara‐Silva, F.; Espinosa‐Matías, S.; Ambrose, B. A.; Vázquez‐Santana, S.; Martínez‐Mena, A.; Márquez‐Guzmán, J.; Martínez, E.; Meyerowitz, E.M.; Álvarez‐Buylla E.R. (2003). Inside‐Out Flowers Characteristic of Lacandonia schismatica Evolved at Least before Its Divergence from a Closely Related Taxon, Triuris brevistylis. International Journal of Plant Sciences. 164: 345-357.
Veselov, D. S., Mustafina, A. R., Sabirjanova, I. B., Akhiyarova, G. R., Dedov, A. V., Veselov, S. U., Kudoyarova, G. R. (2002). Effect of PEG-treatment on the leaf growth response and auxin content in shoots of wheat seedlings. Plant Growth Regul, 38, 191–194.
Vidal, N., barbosa, H., Jacob, S. and Arruda, M. (2015). Comparative study of transgenic and non-transgenic maize (Zea mays) flours commercialized in Brazil, focussing on proteomic analyses. Food Chemistry 180, 288-294.
Villarreal, C., Padilla-Longoria, P. and Alvarez-Buylla, E. R. (2012). General theory of genotype to phenotype mapping: Derivation of epigenetic landscapes from n-node complex gene regulatory networks. Physical Review Letters, 109(11).
Vieten, A., Vanneste, S., Wisniewska, J., Benková, E., Benjamins, R., Beeckman, T., Luschnig, C. and Friml, J. (2005). Functional redundancy of PIN proteins is accompanied by auxin-dependent cross-regulation of PIN expression. Development, 132, 4521-31.
Waddington, C. H. (1942). Canalization of development and the inheritance of acquired characters. Nature, 150, 563-565.
Waddington, C. H. (1953). Genetic assimilation of an acquired character. Evolution, 118-126.
Waddington, C. H. (1957). The Strategy of Genes. London: George Allen y Unwin, Ltd.
Wang, R., Farrona, S., Vincent, C., Joecker, A., Schoof, H., Turcki, F., Alonso-Blanco, C., Coupland, G. and Albani, M. C. (2009). PEP1 regulates perennial flowering in Arabis alpina. Nature, 459, 423-427.
Webber, B., Raghu, S., Edwards, O. (2015). is CRISPR-based gene drive a biocontrol silver bullet or global conservation threat?. PNAS, 112, 10565-10567.
Wegier, A., Piñeyro-Nelson, A, Alarcón, J, Gálvez-Mariscal, A, Alvarez-Buylla, E. R. and Piñero, D. (2011). Recent long-distance transgene flow into wild populations conforms to historical patterns of gene flow in cotton (Gossypium hirsutum) at its centre of origin. Mol Ecol, 20, 4182-4194.
West, G., Inzé, D. and Beemster, G. T. (2004). Cell cycle modulation in the response of the primary root of Arabidopsis to salt stress. Plant Physiol, 135, 1050-8.
Wilkins, A. S. (2008). Waddington’s unfinished critique of neo-Darwinian genetics: then and now. Biological Theory, 3, 224-232.
Wolpert, L., Tickle, C., and Martinez Arias, A. (2015). Principles of Development. Oxford University Press. 652pp.
Wolters, H. and Jürgens, G. (2009). Survival of the flexible: hormonal growth control and adaptation in plant development. Nat Rev Genet, 10, 305-17.
Woodward, A. W. and Bartel, B. (2005). Auxin: regulation, action, and interaction. Ann Bot, 95, 707-35.
World Health Organization. (2014). Global Status Report on Noncommunicable Diseases 2014. World Health Organization. http://doi.org/ISBN 9789241564854.
Wright, S. (1931). Evolution in Mendelian populations. Genetics, 16, 97-159.
Xia, Y., Yu, K., Navarre, D., Seebold, K., Kachroo, A. and Kachroo, P. (2012). The glabra1 mutation affects cuticle formation and plant responses to microbes. Plant Physiol, 154, 833-846.
Yockteng, R., Almeida, A. M. R., Morioka, K., Alvarez-Buylla, E. R. and Specht, C. D. (2013). Molecular evolution and patterns of duplication in the SEP/AGL6-like lineage of the zingiberales: A proposed mechanism for floral diversification. Molecular Biology and Evolution, 30, 2401-2422.