La diversidad de los rizobios nativos de México a la luz de la genómica
Shamayim T. Ramírez-Puebla a, Ernesto Ormeño-Orrillo b, Marco A. Rogel a,
Martha G. López-Guerrero a, Aline López-López c, Julio Martínez-Romero a,
Simoneta Negrete-Yankelevich d y Esperanza Martínez-Romero a, *
a Programa de Ecología Genómica, Centro de Ciencias Genómicas, Universidad Nacional Autónoma de México, Av. Universidad S/N Col. Chamilpa, 62210, Cuernavaca, Morelos, México
b Laboratorio de Ecología Microbiana y Biotecnología, Departamento de Biología, Facultad de Ciencias, Universidad Nacional Agraria La Molina, Av. La Universidad s/n, 526, La Molina, Lima, Perú
c Centro de Investigación en Genética y Ambiente, Universidad Autónoma de Tlaxcala, Km. 10.5 Autopista Tlaxcala-San Martin, 90120 Ixtacuixtla, Tlaxcala, México
d Red de Ecología Funcional, Instituto de Ecología, A.C., Carretera antigua a Coatepec 351, Col. EL Haya, 91070 Coatepec, Veracruz, México
*Autor para correspondencia: esperanzaeriksson@yahoo.com.mx (E. Martínez-Romero)
Resumen
En México hemos identificado nuevas especies de bacterias de los géneros Rhizobium y de Sinorhizobium (ahora llamado oficialmente Ensifer) como R. grahamii, R. mesoamericanum, R. endophyticum, S. mexicanum, S. chiapanecum y S. americanum, además de nuevos linajes de rizobios. El análisis basado en datos genómicos muestra que las especies de Rhizobium cercanas genéticamente comparten significativamente el genoma cromosomal y difieren más en genes contenidos en plásmidos, excepto en algunos plásmidos simbióticos que pueden encontrarse conservados aún en especies no relacionadas que contienen el mismo simbiovar. Los avances en la genómica de rizobios han permitido proponer nuevas especies usando comparaciones genómicas globales y contribuirán al uso más adecuado de estas bacterias como biofertilizantes.
Palabras clave:
Biofertilizantes; Leguminosas; Bacterias; Fijación de nitrógeno
© 2019 Universidad Nacional Autónoma de México, Instituto de Biología. Este es un artículo Open Access bajo la licencia CC BY-NC-ND
Diversity of native Mexican rhizobia under the light of genomics
Abstract
In Mexico we have identified new species of bacteria of the genus Rhizobium and Sinorhizobium (now officially called Ensifer) such as S. mexicanum, S. chiapanecum, S. americanum, R. grahamii, R. mesoamericanum, and R. endophyticum, as well as new rhizobia lineages. Analysis based on genomic data shows that genetically related Rhizobium species significantly share their chromosomal genome but differ in genes contained in plasmids, except in some symbiotic plasmids that can be found even in unrelated species that contain the same simbiovar. Advances in rhizobia genomics have allowed us to propose new species using global genomic comparisons and this knowledge will contribute to the most appropriate use of these bacteria as biofertilizers.
© 2019 Universidad Nacional Autónoma de México, Instituto de Biología. This is an open access article under the CC BY-NC-ND license
Keywords:
Biofertilizers; Leguminous plants; Bacteria; Nitrogen fixation
Introducción
México es un país megadiverso, es el centro de origen de varias especies de leguminosas, algunas de ellas arbóreas y, además, es el centro de domesticación del frijol Phaseolus vulgaris (Bitocchi et al., 2012), que es la leguminosa de mayor consumo humano. En las leguminosas se encuentran bacterias capaces de fijar nitrógeno alojadas en estructuras especializadas llamadas nódulos que se forman en las raíces y, en algunos casos, en los tallos. En aquellos lugares donde una leguminosa es nativa, se encuentran en el suelo las bacterias capaces de establecer simbiosis con dicha planta (Martínez-Romero y Caballero-Mellado, 1996). Estas bacterias, que pertenecen a diversos géneros y que colectivamente son designadas rizobios, ayudan a las plantas a crecer en suelos pobres de nitrógeno, por ello han sido utilizadas como biofertilizantes en la agricultura, permitiendo ahorros en el uso de insumos (Hungria et al., 2000) y la disminución del uso de fertilizantes nitrogenados inorgánicos que contribuyen en la contaminación del suelo y el agua (Savci, 2012). Los rizobios que interactúan con la mayoría de las especies de leguminosas no se han estudiado (López-López, Rosenblueth et al., 2010) y ya que México cuenta con una gran diversidad de plantas, se espera que también exista una gran riqueza bacteriana asociada a éstas. Los estudios genómicos de los rizobios han permitido identificar especies nuevas usando comparaciones genómicas globales, incluyendo los valores de identidad nucleotídica (Konstantinidis y Tiedje, 2005; Ormeño-Orrillo et al., 2015; Richter y Roselló-Móra, 2009) y las filogenias con datos genómicos que permiten una sólida descripción de una especie y la homogenización de los criterios taxonómicos en todas las bacterias.
Rizobios encontrados en México
Una revisión de los patrones de abundancia y distribución en distintos ambientes de los rizobios nativos de México se publicó por Martínez-Romero (2001). La riqueza vegetal de México se traduce en una gran diversidad de bacterias asociadas a plantas y aquí revisaremos ejemplos de rizobios encontrados en México, algunos con distribución aparentemente global y otros endémicos. Describimos a Rhizobium etli oriundo de México (Segovia et al., 1993). Esta especie se encuentra también en otras regiones geográficas donde el frijol ha sido introducido (Aserse et al., 2012; Cao et al., 2014; Martínez-Romero, 2003; Mhamdi et al., 1999; Robledo et al., 2011). Inicialmente, la diversidad de R. etli se estudió con patrones electroforéticos de enzimas metabólicas y la alta diversidad encontrada se atribuyó a su antigüedad, además se discutió que R. etli podría abarcar a distintas especies (Piñero et al., 1988). Posteriormente, se ha identificado una diversidad amplia de linajes de rizobios de los nódulos de Phaseolus vulgaris con análisis tanto moleculares (Miranda-Sánchez et al., 2016; Ribeiro et al. 2013; Wang et al., 2011) como genómicos en distintas regiones de México, del Caribe (Carrascal et al., 2016; Díaz-Alcántara et al., 2014; Santamaría et al., 2017; Tong et al., 2018) y de China, donde el frijol fue introducido y es cultivado a gran escala (Cao et al., 2014). Se han reclasificado a algunas cepas previamente consideradas como R. etli en otras especies cercanas, R. phaseoli (Ramírez-Bahena et al., 2008), R. ecuadorense (Ribeiro et al., 2015) y R. esperanzae (Cordeiro et al., 2017), mientras otros linajes nuevos no han sido todavía nombrados como nuevas especies (López-Guerrero et al., 2012). Es notable que distintas especies de rizobios con capacidad de formar nódulos en P. vulgaris comparten un plásmido simbiótico conservado que representa al simbiovar phaseoli o tropici, lo que indica que los plásmidos pueden transferirse entre rizobios en condiciones naturales (Cao et al., 2014), y se ha confirmado en experimentos de laboratorio (Brom et al., 1988; Martínez et al., 1987; Rogel et al., 2001). Es importante aclarar que el término simbiovar sustituyó al de biovar y refleja la especificidad del rizobio por la planta (Rogel et al., 2011). En R. etli descubrimos un nuevo simbiovar, mimosae (Wang et al., 1999) que tiene la capacidad de nodular mimosas y frijol; su genoma ha sido ya reportado (Rogel et al., 2014).
En México, además de P. vulgaris, se cultiva P. lunatus (ibe en México, frijol lima en inglés) que normalmente se asocia con cepas de Bradyrhizobium. Algunas de las cepas de bradyrizobios nativas de la selva de Los Tuxtlas en México son los simbiontes de P. lunatus (López-López et al., 2013), mientras que en Perú se identificaron especies diferentes (Durán et al., 2014; Ormeño-Orrillo et al., 2006; Ormeño-Orrillo, Rey, Durán, Canchaya, Rogel et al., 2017; Ormeño-Orrillo, Rey, Durán, Canchaya, Zúñiga-Dávila et al., 2017). Estos resultados indican que las bacterias que nodulan a P. lunatus no tienen una distribución global y son endémicas de cada región. Los simbiontes de distintas especies de Phaseolus (Parker, 2002), incluyendo P. microcarpus (Servín-Garcidueñas et al., 2016), pertenecen al género Bradyrhizobium (Servín-Garcidueñas et al., 2014).
Para explicar la evolución de los distintos simbiontes de Phaseolus, presentamos distintas evidencias que apoyan la ocurrencia de un cambio reciente de simbionte hacia bacterias del género Rhizobium en P. vulgaris y especies relacionadas (Servín-Garcidueñas et al., 2014). Propusimos además un esquema de evolución rápida de rizobios en campos agrícolas que conduciría a la generación de diversidad de cepas de una misma especie (Martínez et al., 2016).
Rhizobium tropici (Martínez-Romero et al., 1991) y Rhizobium leucaenae (Gamez-Reyes et al., 2017; Ribeiro et al., 2012) son rizobios presentes en México, pero también distribuidos ampliamente por lo menos en regiones tropicales de América y África. Los hemos encontrado en nódulos de árboles de Gliricidia sepium, la “alfalfa de los trópicos” en Veracruz (Acosta-Durán y Martínez-Romero, 2002). El genoma de R. tropici y de bacterias relacionadas permitió comenzar a entender las bases genéticas de su amplio rango de hospederos y de su tolerancia al estrés (Ormeño-Orrillo, Menna et al., 2012) y además, fue la base para definir nuevas especies cercanas a R. tropici (Dall’Agnol et al., 2013; Ribeiro et al., 2012) que comparten plásmidos simbióticos y por tanto el mismo simbiovar, designado tropici.
Rhizobium gallicum es otro rizobio asociado a frijol (Amarger et al., 1997). A pesar de su nombre, gallicum, que significa “de Galia” (nombre romano de Francia), esta especie parece ser otro simbionte originario de México que fue dispersado por semillas hacia Europa. En México lo encontramos en una minoría de nódulos de frijol, pero es el principal simbionte del ayocote (Phaseolus coccineus), otra planta domesticada en América (Silva et al., 2003). Los genomas de algunas cepas de R. gallicum se han publicado recientemente (Bustos et al., 2017).
Hemos descrito nuevas especies de rizobios con capacidad de nodular diversas leguminosas, R. grahamii (Althabegoiti et al., 2014; Peralta et al., 2016), R. mesoamericanum (López-López et al., 2012), R. mayense, R. jaguaris y R. calliandrae (Rincón-Rosales et al., 2013) y una especie que no nodula, pero se encuentra dentro de las semillas de frijol, R. endophyticum (López-López, Rogel et al., 2010). También hay rizobios simbióticos que se encuentran sobre la superficie de semillas de Phaseolus vulgaris (Pérez-Ramírez et al., 1998). Suponemos que R. etli, y quizás también algunas otras especies han sido acarreadas con las semillas de frijol transportadas por el ser humano. Las bacterias en semillas contribuirían con su dispersión fuera de su sitio de origen (Cao et al., 2014). Se han reportado R. grahamii, R. trifolii, R. phaseoli y Sinorhizobium americanum en semillas del frijol (Peralta et al., 2016).
Aislamos 2 nuevas especies de Sinorhizobium de una leguminosa oriunda de Chiapas y Morelos, Acaciella angustissima relacionada con las acacias (Lloret et al., 2007; Rincón-Rosales et al., 2009). Estas bacterias están emparentadas con las especies que se encuentran en nódulos de acacias en África (Lorquin et al., 1997). Describimos otra especie, Sinorhizobium americanum aislado de varias especies de Acacia en la Sierra de Huautla en Morelos (Toledo et al., 2003). Hasta el momento, estas especies: S. mexicanum, S. chiapanecum y S. americanum, junto con R. grahamii, R. endophyticum, R. calliandrae, R. jaguaris y R. mayense, son los rizobios que solo se han encontrado en México y que representarían endemismos.
Otros autores han aislado cepas de Bradyrhizobium de nódulos de algunas especies silvestres de Phaseolus, Desmodium y Macroptilium que crecen en Chihuahua, México (Parker, 2002). Los análisis realizados no permitieron discernir la especie a la que pertenecían, pero se obtuvieron representantes de los 2 grandes grupos en los que se suele dividir a Bradyrhizobium, el grupo aliado a B. japonicum y el aliado a B. elkanii. Encontramos una alta diversidad de cepas de Bradyrhizobium en un estudio de los simbiontes de 5 especies mexicanas de Lupinus de Morelos. El análisis de la secuencia del gen ribosomal 16S de 3 cepas representativas las ubicó en el grupo aliado a B. japonicum (Barrera et al., 1997).
Hemos aislado distintos rizobios y sinorizobios y en pocos casos burkholderias de mimosas en distintas regiones de México (Bontemps et al., 2016; Ormeño-Orrillo, Rogel, Chueire et al., 2012). Estos datos apoyan una distribución de especies distinta en México y en Brasil ya que allí las bacterias simbiontes de mimosas son predominantemente burkholderias y ésto se ha atribuido a los niveles de nitrógeno más bajos que existen en Brasil y que permiten la nodulación de las burkholderias, la cual fácilmente se inhibe con la presencia de nitrógeno (Elliot et al., 2009).
Descubrimos 27 nuevos linajes de Bradyrhizobium en un estudio sobre los efectos de la tala de la selva tropical y del cambio de uso del suelo en el bosque tropical de Los Tuxtlas, México (Ormeño-Orrillo, Rogel, Lloret et al., 2012). Tres de ellos son semejantes a otros descritos por Parker (2004) como simbiontes de árboles tropicales en Centroamérica, mientras que otros 24 linajes no tienen parientes reportados en otras partes del mundo, por lo que podrían ser endémicos de México. En el bosque de Los Tuxtlas encontramos además: R. etli, R. leucaenae, R. mesoamericanum, R. hainanense, S. americanum, M. amorphae y Burkholderia tuberum, más 3 nuevos linajes de Rhizobium sin parientes reportados en otros lugares (Ormeño-Orrillo et al., 2009; Ormeño-Orrillo, Rogel, Lloret et al., 2012). También en Veracruz, pero en campos de agricultores en Perote, encontramos en los nódulos de Phaseolus dumosus cepas cercanas a R. etli, R. leguminosarum y un nuevo linaje de Rhizobium que describimos con datos genómicos (Ramírez-Puebla et al., 2018). Estos rizobios muestran distintas capacidades simbióticas, lo que indica que tal vez se podría mejorar la fijación de nitrógeno en frijol usando las cepas más eficientes como inoculantes.
En los nódulos de las leguminosas de la especie Lotus oroboides se encontraron rizobios de las especies Mesorhizobium huakuii y Mesorhizobium loti en la sierra Madre Occidental de Chihuahua, México (Qian y Parker, 2002). Las especies M. huakuii y M. loti fueron descritas inicialmente a partir de bacterias aisladas en Asia. Hemos encontrado otras 2 especies de Mesorhizobium originalmente descritas en Brasil, Senegal, Sudán y en China, Mesorhizobium amorphae y Mesorhizobium plurifarium en Morelos (Wang et al., 2003) y en la selva de Los Tuxtlas, Veracruz (Ormeño-Orrillo et al., 2009). En forma similar se encontró en una leguminosa tropical (Sesbania herbacea) una nueva especie de Rhizobium, a la cual designamos R. huautlense por ser originario de la sierra de Huautla en Morelos (Wang et al., 1998); posteriormente, la misma especie fue encontrada en Venezuela nodulando otras especies de Sesbania (Vinuesa et al., 2005).
En un caso diferente, los rizobios encontrados en México no son nativos y fueron probablemente introducidos junto con los cultivos de alfalfa (Medicago sativa) y de otra leguminosa forrajera (Medicago lupulina) (Silva et al., 2007). Estas bacterias aisladas de nódulos obtenidos en Guanajuato, Estado de México y Morelos pertenecen a las especies S. meliloti y S. medicago, cuyo sitio de origen es Asia Central. Es posible que S. meliloti y S. medicae llegaran con las semillas de alfalfa o que fueran introducidas con suelo o como inoculantes.
Rizobios en plantas no leguminosas de México
Es de especial interés que los rizobios también colonicen abundantemente plantas no leguminosas de importancia agrícola y que puedan promover su crecimiento como lo hacen las bacterias conocidas como PGPR (siglas en inglés de rizobacterias promotoras del crecimiento vegetal) (Friesen et al., 2011). En México, descubrimos que R. etli coloniza la rizósfera y el interior del maíz y actúa como PGPR (Gutíerrez-Zamora y Martínez-Romero, 2001). Además, se ha demostrado que R. etli inhibe la proliferación de patógenos en plantas de papa y maíz (Reiter et al., 2002; Rosenblueth y Martínez-Romero, 2004). Obtuvimos una nueva especie no nodulante de Rhizobium de las raíces de agaves en Chiapas con propiedades de promoción del crecimiento vegetal (Ruiz-Valdiviezo et al., 2017).
Las bases moleculares y genéticas de las interacciones de rizobios con plantas no leguminosas han sido menos estudiadas que con plantas leguminosas (Rosenblueth y Martínez-Romero, 2006). Las capacidades de rizobios para utilizar diversas fuentes de carbono parecen ser muy extensas y probablemente contribuyen para la colonización de diversas plantas (López-Guerrero et al., 2013). Seguramente los genes necesarios para el transporte y metabolismo de diversos compuestos naturales producidos por las plantas solo se expresan en presencia de éstos (López-Guerrero et al., 2013), lo que significaría que el potencial metabólico de estas bacterias no se expresa en vida libre y no puede ser fácilmente estudiado.
Diversidad a la luz de la genómica
Los enfoques moleculares mediante la secuenciación de genes clave que se designan marcadores moleculares (fig. 1) permiten una adecuada clasificación de las bacterias (Alexandre et al., 2008; Case et al., 2007; Hernández-Lucas et al., 2004; Martínez-Romero et al., 2010) y además, permiten comenzar a entender sus relaciones evolutivas. La clasificación de rizobios basada en pruebas fenotípicas ha sido fuertemente criticada, ya que este tipo de análisis puede subestimar las diferencias intrínsecas de las especies (Ormeño-Orrillo y Martínez-Romero, 2013). Un conocimiento más completo deriva de estudios genómicos en los que se determinan las secuencias de todos los genes y además de las regiones intergénicas (de Lajudie y Martínez-Romero, 2017; Galibert et al., 2001; González et al., 2006; Young et al., 2006). Estos estudios permiten establecer relaciones filogenéticas con miles de genes en vez de con unos pocos. Algunas conclusiones generales derivadas de estudios genómicos muestran que las filogenias descritas con pocos genes son muy semejantes a las obtenidas con miles de genes, pero en ocasiones, genes únicos dan filogenias discordantes (López-Guerrero et al., 2012; Ormeño-Orrillo et al., 2015). Los genomas de cepas diversas de una especie han revelado la existencia del pangenoma, que es el conjunto de genes presentes en toda la especie repartido entre distintas cepas. En algunos casos, una decena de cepas puede cubrir el pangenoma. En otros casos, el pangenoma es abierto y cada vez que se obtiene una nueva secuencia genómica se aportan nuevos genes al pangenoma. Los rizobios poseen pangenomas grandes, tal vez con relación al alto contenido de genes de plásmidos que pueden ser transferidos entre especies.
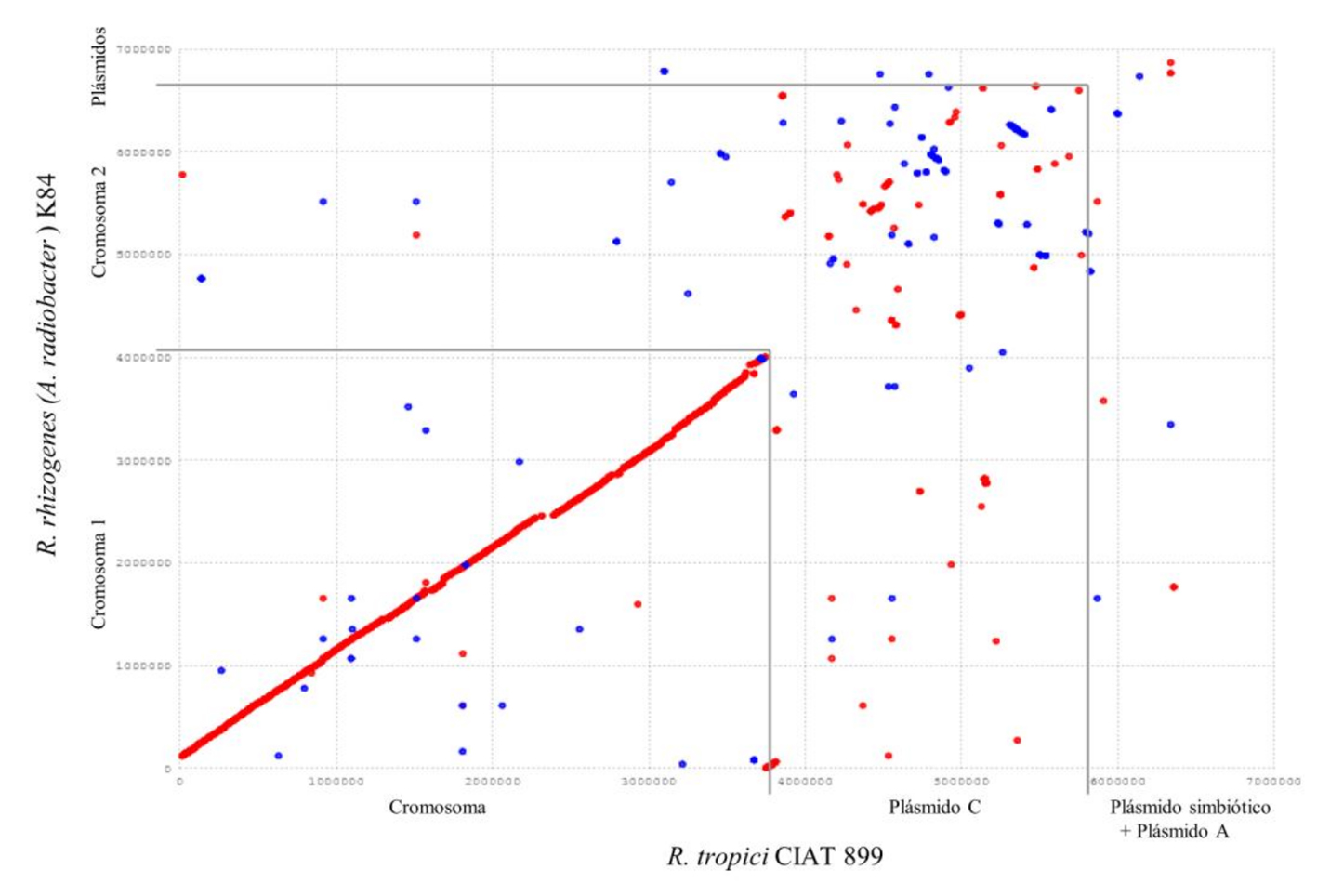
Las secuencias completas de los genomas han permitido reconocer que existe en cada especie un conjunto de genes comunes con conservación promedio de las secuencias nucleotídicas, arriba del 94% (Goris et al., 2007; Konstantinidis y Tiedje, 2005). Cabe mencionar que muchos de estos genes conservados mantienen la sintenia (fig. 2), es decir, tienen un orden parecido en el cromosoma (Crossman et al., 2008; Guerrero et al., 2005). Los estudios genómicos de cepas de rizobios muestran que muchos genes conservados se encuentran básicamente en el cromosoma (fig. 2) y en unos pocos crómidos (Harrison et al., 2010), y que las diferencias más grandes se encuentran principalmente en varios plásmidos (Althabegoiti et al., 2014). Una característica sobresaliente de cepas de Rhizobium, Sinorhizobium y Agrobacterium es que gran parte del genoma se encuentra codificado en plásmidos (López-Guerrero et al., 2012).
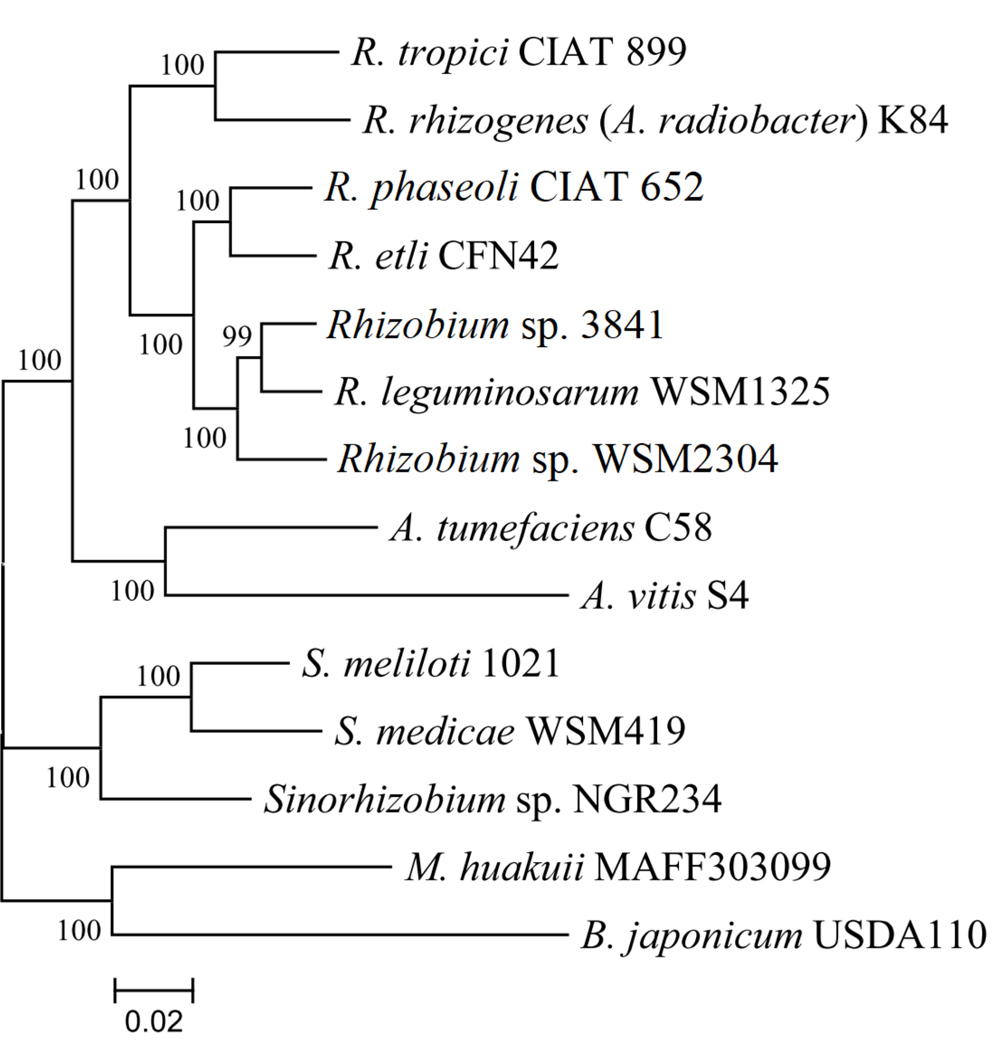
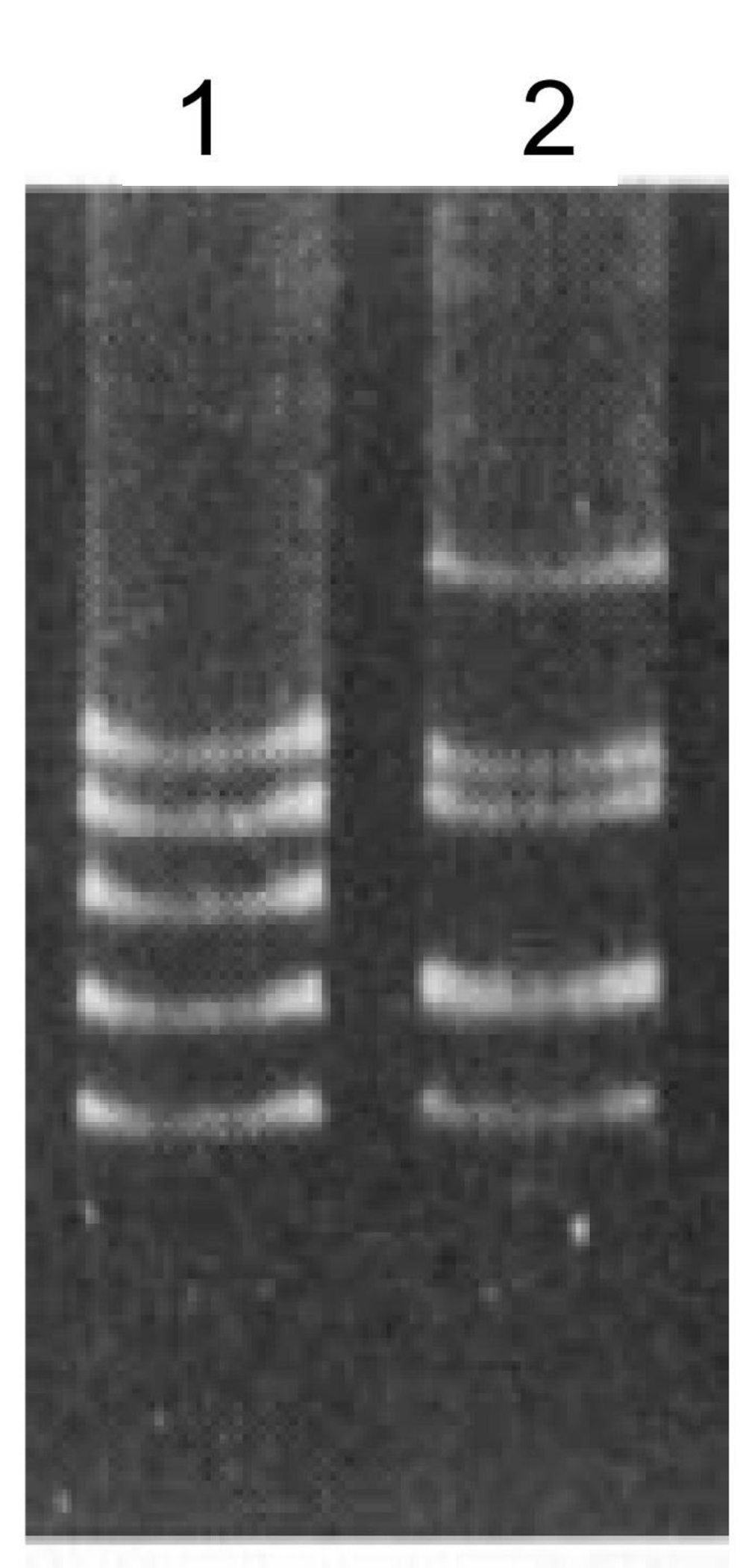
Entonces, cuando estudiamos diversidad, estudiamos principalmente diversidad de plásmidos, ¿qué tanto son éstos responsables de la especiación? ¿Qué hace única a una especie? ¿Cuánto tiempo se requiere para que un proceso de especiación concluya con la génesis de una especie nueva? Aún no tenemos buenas respuestas para estas preguntas. Sin embargo, sí se cuentan con reglas publicadas de taxonomía para la definición de especies en rizobios (Graham et al., 1991) que durante muchos años han considerado enfoques polifásicos que incluyen características fenotípicas y genotípicas. En éstas últimas, ahora se recomiendan los análisis de genomas. Las características fenotípicas, que se exigen, han sido ampliamente criticadas por su pobre valor en reconocer y delinear especies (Ormeño-Orrillo y Martínez-Romero, 2013). Pueden ocurrir diversos rearreglos genómicos y cambios muy significativos en los contenidos plasmídicos y de islas genómicas en tiempos evolutivos cortos, mientras las secuencias de genes conservados cambian limitadamente (Martínez et al., 2016). Aún entre cepas cercanas, los patrones de plásmidos pueden variar (fig. 3; Wang et al., 1999). La adquisición o pérdida de un plásmido puede cambiar drásticamente la adaptación a un nicho completamente diferente (evolución en un paso); ésto se ilustra significativamente con rizobios y cepas relacionadas (Martínez-Romero, 2009). R. tropici forma nódulos, mientras que una especie cercana, R. rhizogenes, produce una proliferación de raíces debido al plásmido Ri. Las relaciones filogenéticas de estas cepas utilizando un gen marcador dnaK se muestra en la figura 1. En los rizobios, los determinantes simbióticos se encuentran en plásmidos que también codifican transportadores de diversos compuestos y enzimas para síntesis de vitaminas (García-de los Santos et al., 1996; López-Guerrero et al., 2012). La presencia o ausencia de plásmidos y/o de islas genómicas, algunas de ellas tan grandes como plásmidos, puede cambiar las características fenotípicas y por supuesto los valores de hibridación ADN-ADN, que tanta importancia tienen en taxonomía.
Si una especie se distingue por sus capacidades fenotípicas únicas (Richter y Roselló-Mora, 2009) y en rizobios éstas parecen estar determinadas en gran medida por plásmidos o islas genómicas, entonces la regla de que las especies se deben definir por secuencias cromosomales y no por accesorias, no se cumpliría. Si se requieren 50 millones de años para cambiar 1% la secuencia de un gen 16S ribosomal, en este período hay tiempo más que suficiente para rearreglos genómicos, eventos de transferencia de plásmidos y cambios significativos en el contenido de genes (Martínez et al., 2016). En rizobios, ésto sería probablemente la base de la especiación; entonces, la riqueza de rizobios de México se podría traducir a conjuntos de genes nuevos y exitosos para la interacción con plantas leguminosas y no leguminosas o para la resistencia a estrés ambiental o para muchas otras funciones como degradación de contaminantes (González-Paredes et al., 2013) o producción de antibióticos y otros compuestos que no conocemos aún. ¿Cómo se puede proteger entonces la diversidad en estos términos?
Seguramente aún existe una gran diversidad de rizobios por explorar en México. La riqueza vegetal de México ha propiciado una gran diversidad de rizobios que con certeza pueden ser considerados nativos de México. Indudablemente, su preservación requiere de la conservación de los hábitats naturales de este país. Los genes para la producción de compuestos novedosos o para la colonización de la rizósfera o de las raíces del maíz, podrían utilizarse para aplicaciones biotecnológicas. El análisis de cuantiosos genomas disponibles de muy diversos rizobios nos dará seguramente valiosas respuestas, aunque la enorme cantidad de genes hipotéticos con función desconocida representan un obstáculo en esta vía.
Agradecimientos
S. negrete Yankelevich agradece al Conacyt por el proyecto “Biodiversidad en la milpa y su suelo: bases para la seguridad alimentaria de mujeres y niños en el Cofre de Perote” Problemas Nacionales 246999 y E. Martínez Romero a PAPIIT UNAM IN207718.
Referencias
Acosta-Durán, C. y Martínez-Romero, E. (2002). Diversity of rhizobia from nodules of the leguminous tree Gliricidia sepium, a natural host of Rhizobium tropici. Archives of Microbiology, 178, 161–164.
Alexandre, A., Laranjo, M., Young, J. P. W. y Oliveira, S. (2008). dnaJ is a useful phylogenetic marker for alphaproteobacteria. International Journal of Systematic and Evolutionary Microbiology, 58, 2839–2849.
Althabegoiti, M. J., Ormeño-Orrillo, E., Lozano, L., Torres-Tejerizo, G., Rogel, M. A., Mora, J. et al. (2014). Characterization of Rhizobium grahamii extrachromosomal replicons and their transfer among rhizobia. BMC Microbiology, 14, 6.
Amarger, N., Macheret, V. y Laguerre, G. (1997). Rhizobium gallicum sp. nov. and Rhizobium giardinii sp. nov., from Phaseolus vulgaris nodules. International Journal of Systematic Bacteriology, 47, 996–1006.
Aserse, A. A., Räsänen, L. A., Assefa, F., Hailemariam, A. y Lindström, K. (2012). Phylogeny and genetic diversity of native rhizobia nodulating common bean (Phaseolus vulgaris L.) in Ethiopia. Systematic and Applied Microbiology, 35, 120–131.
Barrera, L. L., Trujillo, M. E., Goodfellow, M., García, F. J., Hernández-Lucas, I., Dávila, G. et al. (1997). Biodiversity of bradyrhizobia nodulating Lupinus spp. 358. International Journal of Systematic Bacteriology, 47, 1086–1091.
Bitocchi, E., Nanni, L., Bellucci, E., Rossi, M., Giardini, A., Zeuli, P. S. et al. (2012). Mesoamerican origin of the common bean (Phaseolus vulgaris L.) is revealed by sequence data. Proceedings of the National Academy of Sciences, USA, 109, E788–E796.
Bontemps, C., Rogel, M. A., Wiechmann, A., Mussabekova, A., Moody, S., Simon, M. F. et al. (2016). Endemic Mimosa species from Mexico prefer alphaproteobacterial rhizobial symbionts. New Phytologist, 209, 319–333.
Brom, S., Martínez, E., Dávila, G. y Palacios, R. (1988). Narrow- and broad-host-range symbiotic plasmids of Rhizobium spp. strains that nodulate Phaseolus vulgaris. Applied and Environmental Microbiology, 54, 1280–1283.
Bustos, P., Santamaría, R. I., Pérez-Carrascal, O. M., Acosta, J. L., Lozano, L., Juárez, S. et al. (2017). Complete genome sequences of three Rhizobium gallicum symbionts associated with common bean (Phaseolus vulgaris). Genome Announcements, 5, e00030-17.
Cao, Y., Wang, E., Zhao, L., Chen, W. M. y Wei, G. H. (2014). Diversity and distribution of rhizobia nodulated with Phaseolus vulgaris in two ecoregions of China. Soil Biology and Biochemistry, 78, 128–137.
Carrascal, O. M. P., Juárez, S., Polz, M. F., Vinuesa, P. y González, V. (2016). Population genomics of the symbiotic plasmids of sympatric nitrogen-fixing Rhizobium species associated with Phaseolus vulgaris. Environmental Microbiology, 18, 2660–2676.
Case, R. J., Boucher, Y., Dahlloef, I., Holmstroem, C., Doolittle, W. F. y Kjelleberg, S. (2007). Use of 16S rRNA and rpoB genes as molecular markers for microbial ecology studies. Applied and Environmental Microbiology, 73, 278–288.
Cordeiro, A. B., Ribeiro, R. A., Helene, L. C. F. y Hungria, M. (2017). Rhizobium esperanzae sp. nov., a N2-fixing root symbiont of Phaseolus vulgaris from Mexican soils. International Journal of Systematic Evolutionary Microbiology, 67, 3937–3945.
Crossman, L. C., Castillo-Ramírez, S., Mcannula, C., Lozano, L., Vernikos, G. S., Acosta, J. L. et al. (2008). A common genomic framework for a diverse assembly of plasmids in the symbiotic nitrogen fixing bacteria. Plos One, 3, e2567.
Dall’Agnol, R. F., Ribeiro, R. A., Ormeño-Orrillo, E., Rogel, M. A., Delamuta, J. R., Andrade, D. S. et al. (2013). Rhizobium freirei sp. nov., a symbiont of Phaseolus vulgaris that is very effective at fixing nitrogen. International Journal of Systematic and Evolutionary Microbiology, 63, 4167–4173.
de Lajudie, P. y Martínez-Romero, E. (2017). International Committee on Systematics of Prokaryotes Subcommittee on the taxonomy of Agrobacterium and Rhizobium Minutes. International Journal of Systematic and Evolutionary Microbiology, 67, 516–520.
Díaz-Alcántara, C. A., Ramírez-Bahena, M. H., Mulas, D., García-Fraile, P., Gómez-Moriano, A., Peix, A. et al. (2014). Analysis of rhizobial strains nodulating Phaseolus vulgaris from Hispaniola Island, a geographic bridge between Meso and South America and the first historical link with Europe. Systematic and Applied Microbiology, 37, 149–156.
Durán, D., Rey, L., Mayo, J., Zúñiga-Dávila, D., Imperial, J., Ruiz-Argüeso, T. et al. (2014). Bradyrhizobium paxllaeri sp. nov. and Bradyrhizobium icense sp. nov., nitrogen-fixing rhizobial symbionts of Lima bean (Phaseolus lunatus L.) in Peru. International Journal of Systematic and Evolutionary Microbiology, 64, 2072–2078.
Elliott, G. N, Chou, J. H., Chen, W. M., Bloemberg, G. V., Bontemps, C., Martínez-Romero, E. et al. (2009). Burkholderia spp. are the most competitive symbionts of Mimosa, particularly under N-limited conditions. Environmental Microbiology, 11, 762–778.
Friesen, M. L., Porter, S. L., Stark, S. C., von Wettberg, E. J., Sachs, J. L. y Martínez-Romero, E. (2011). Microbially mediated plant functional traits. Annual Review of Ecology Evolution and Systematics, 42, 23–46.
Galibert, F., Finan, T. M., Long, S. R., Puhler, A., Abola, P., Ampe, F. et al. (2001). The composite genome of the legume symbiont Sinorhizobium meliloti. Science, 293, 668–672.
Gamez-Reyes, A., Becerra-Lobato, N., Ramírez-Trujillo, J. A., Martínez-Romero, E., Dunn, M. F. y Hernandez-Lucas, I. (2017). The Rhizobium leucaenae CFN 299 pSym plasmid contains genes expressed in free life and symbiosis, as well as two replication systems. Annals of Microbiology, 67, 263–273.
García-de Los Santos, A., Brom, S. y Romero, D. (1996). Rhizobium plasmids in bacteria-legume interactions. World Journal of Microbiology and Biotechnology, 12, 119–125.
González, V., Acosta, J. L., Santamaría, R. I., Bustos, P., Fernández, J. L., Hernández-González, I. L. et al. (2010). Conserved symbiotic plasmid DNA sequences in the multireplicon pangenomic structure of Rhizobium etli. Applied Environmental Microbiology, 76, 1604-1614.
González, V, Santamaría, R. I., Bustos, P., Hernández-González, I., Medrano-Soto, A., Moreno-Hagelsieb, G. et al. (2006). The partitioned Rhizobium etli genome: genetic and metabolic redundancy in seven interacting replicons. Proceedings of the National Academy of Sciences, USA, 103, 3834–3839.
González-Paredes, Y., Alarcón, A., Ferrera-Cerrato, R., Almaraz, J. J., Martínez-Romero, E., Cruz-Sánchez, J. S. et al. (2013). Tolerance, growth and degradation of phenanthrene and benzo[α]pyrene by Rhizobium tropici CIAT 899 in liquid culture medium. Applied Soil Ecology, 63, 105–111.
Goris, J., Konstantinidis, K. T., Klappenbach, J. A., Coenye, T., Vandamme, P. y Tiedje, J. M., (2007). DNA-DNA hybridization values and their relationship to whole-genome sequence similarities. International Journal of Systematic and Evolutionary Microbiology, 57, 81–91.
Graham, P. H., Sadowsky, M. J., Keyser, H. H., Barnet, Y. M., Bradley, R. S., Cooper, J. E. et al. (1991). Proposed minimal standards for the description of new genera and species of root-and stem-nodulating bacteria. International Journal of Systematic Bacteriology, 41, 582–587.
Guerrero, G., Peralta, H., Aguilar, A., Díaz, R., Villalobos, M.A., Medrano-Soto, A. et al. (2005). Evolutionary, structural and functional relationships revealed by comparative analysis of syntenic genes in Rhizobiales. BMC Developmental Biology, 5, 55.
Gutiérrez-Zamora, M. L. y Martínez-Romero, E. (2001). Natural endophytic association between Rhizobium etli and maize (Zea mays L.). Journal of Biotechnology, 91, 117–126.
Harrison, P. W., Lower, R. P. J., Kim, N. K. D. y Young, J. W. (2010). Introducing the bacterial chromid: not a chromosome, not a plasmid. Trends in Microbiology, 18, 141–148.
Hernández-Lucas, I., Rogel-Hernández, M. A., Segovia, L., Rojas-Jiménez, K. y Martínez-Romero, E. (2004). Phylogenetic relationships of rhizobia based on citrate synthase gene sequences. Systematic and Applied Microbiology, 27, 703–706.
Hungría, M., Vargas, M. A. T., Campo, R. J., Chueire, L. M. O. y Andrade, D. S. (2000). The Brazilian experience with the soybean (Glycine max) and common bean (Phaseolus vulgaris) symbioses. En F. O. Pedrosa, M. Hungria, G. Yates y W. E. Newton (Eds.), Nitrogen fixation: from molecules to crop production. Países Bajos: Kluwer Academic Publishers.
Konstantinidis, K. T. y Tiedje, J. M. (2005). Genomic insights that advance the species definition for prokaryotes. Proceedings of the National Academy of Sciences, USA, 102, 2567–2572.
Lloret, L., Ormeño-Orrillo, E., Rincón, R., Martínez-Romero, J., Rogel-Hernández, M.A. y Martínez-Romero, E. (2007). Ensifer mexicanus sp. nov. a new species nodulating Acacia angustissima (Mill.) Kuntze in Mexico. Systematic and Applied Microbiology, 30, 280–290.
López-Guerrero, M. G., Ormeño-Orrillo, E., Acosta, J. L., Mendoza-Vargas, A., Rogel, M. A., Ramírez, M. A. et al. (2012). Rhizobial extrachromosomal replicon variability, stability and expression in natural niches. Plasmid, 68, 149–158.
López-Guerrero, M. G., Ormeño-Orrillo, E., Rosenblueth, M., Martínez-Romero, J. y Martínez-Romero, E. (2013). Buffet hypothesis for microbial nutrition at the rhizosphere. Frontiers in Plant Sciences, 4, 1–4.
López-López, A., Negrete-Yankelevich, S., Rogel, M.A., Ormeño-Orrillo, E., Martínez, J. y Martínez-Romero, E. (2013). Native bradyrhizobia from Los Tuxtlas in Mexico are symbionts of Phaseolus lunatus (Lima bean). Systematic and Applied Microbiology, 36, 33–38.
López-López, A., Rogel, M. A., Barois, I., Ortiz-Ceballos, A. I., Martínez, J., Ormeño-Orrillo, E. et al. (2012). Rhizobium grahamii sp. nov. from Dalea leporina, Leucaena leucocephala, Clitoria ternatea nodules, and Rhizobium mesoamericanum sp. nov. from Phaseolus vulgaris, siratro, cowpea and Mimosa pudica nodules. International Journal of Systematic and Evolutionary Microbiology, 62, 2264–2271.
López-López, A., Rogel, M. A., Ormeño-Orrillo, E., Martínez-Romero, J. y Martínez-Romero, E. (2010). Phaseolus vulgaris seed-borne endophytic community with novel bacterial species such as Rhizobium endophyticum sp. nov. Systematic Applied Microbiology, 33, 322–327.
López-López, A., Rosenblueth, M., Martínez, J. y Martínez-Romero, E. (2010). Rhizobial symbioses in tropical legumes and non-legumes. En P. Dion (Ed.), Soil biology and agriculture in the tropics (pp.163–184). Quebec City: Springer.
Lorquin, J., Lortet, G., Ferro, M., Mear, N., Prome, J. C. y Boivin, C. (1997). Sinorhizobium teranga bv. acaciae ORS1073 and Rhizobium sp. strain ORS1001, two distantly related Acacia-nodulating strains, produce similar nod factors that are O carbamoylated, N methylated, and mainly sulfated. Journal of Bacteriology, 179, 3079–3083.
Martínez, J., Negrete-Yankelevich, S., Gómez-Godinez, L., Reyes, J., Degli-Esposti, M. y Martínez-Romero, E. (2016). Short-term evolution of rhizobial strains towards sustainability in agriculture. En S. Castro-Sowinski (Ed.), Microbial models: from environment to industrial sustainability. Microorganims for sustainability, Vol. I. Singapore: Springer.
Martínez, E., Palacios, R. y Sánchez, F. (1987). Nitrogen-fixing nodules induced by Agrobacterium tumefaciens harboring Rhizobium phaseoli plasmids. Journal of Bacteriology, 169, 2828–2834.
Martínez-Romero, E. (2001). Poblaciones de rhizobias nativas de México. Acta Zoológica Mexicana. Número especial, 1, 29–38.
Martínez-Romero, E. (2003). Diversity of Rhizobium-Phaseolus vulgaris symbiosis: overview and perspectives. Plant and Soil, 252, 11–23.
Martínez-Romero, E. (2009). Coevolution in Rhizobium-legume symbiosis? DNA and Cell Biology, 28, 361–370.
Martínez-Romero, E. y Caballero-Mellado, J. (1996). Rhizobium phylogenies and bacterial genetic diversity. Critical Reviews in Plant Sciences, 15, 113–140.
Martínez-Romero, J., Ormeño-Orrillo, E., Rogel, M. A., López-López, A. y Martínez-Romero, E. (2010). Trends in rhizobial evolution and some taxonomic remarks. En P. Pontarotti (Ed.), Evolutionary biology – concepts, molecular and morphological evolution (pp.301–315). New York: Springer.
Martínez-Romero, E., Segovia, L., Martins-Mercante, F., Antonio-Franco, A., Graham, P. y Aurelio-Pardo, M. (1991). Rhizobium tropici, a novel species nodulating Phaseolus vulgaris L. beans and Leucaena sp. trees. International Journal of Systematic Bacteriology, 41, 417–426.
Mhamdi, R., Jebara, M., Aouani, M. E., Ghrir, R. y Mars, M. (1999). Genotypic diversity and symbiotic effectiveness of rhizobia isolated from root nodules of Phaseolus vulgaris L. grown in Tunisian soils. Biology and Fertility of Soils, 28, 13–320.
Miranda-Sánchez, F., Rivera, J. y Vinuesa, P. (2016). Diversity patterns of Rhizobiaceae communities inhabiting soils, root surfaces and nodules reveal a strong selection of rhizobial partners by legumes. Environmental Microbiology, 18, 2375–2391.
Ormeño-Orrillo, E. y Martínez-Romero, E. (2013). Phenotypic tests in Rhizobium species description: an opinion and (a sympatric speciation) hypothesis. Systematic and Applied Microbiology, 36, 145–147.
Ormeño-Orrillo, E., Menna, P., Almeida, L. G., Ollero, F. J., Nicolás, M. F., Pains-Rodrigues, E. (2012). Genomic basis of broad host range and environmental adaptability of Rhizobium tropici CIAT 899 and Rhizobium sp. PRF 81 which are used in inoculants for common bean (Phaseolus vulgaris L.). BMC Genomics, 13, 735.
Ormeño-Orrillo, E., Rey, L., Durán, D., Canchaya, C. A., Rogel, M. A., Zúñiga-Dávila, D. et al. (2017). Draft genome sequence of Bradyrhizobium paxllaeri LMTR 21T isolated from Lima bean (Phaseolus lunatus) in Peru. Genomics Data, 13, 38–40.
Ormeño-Orrillo, E., Rey, L., Durán, D., Canchaya, C. A., Zúñiga-Dávila, D., Imperial, J. et al. (2017). Genome sequence of Bradyrhizobium sp. LMTR 3, a diazotrophic symbiont of Lima bean (Phaseolus lunatus). Genomics Data, 13, 35–37.
Ormeño-Orrillo, E., Rogel, M. A., Chueire, L. M., Tiedje, J. M., Martínez-Romero, E. y Hungria, M. (2012). Genome sequences of Burkholderia sp. strains CCGE1002 and H160, isolated from legume nodules in Mexico and Brazil. Journal of Bacteriology, 194, 6927.
Ormeño-Orrillo, E., Rogel, M. A., Lloret, L., López-López, A., Martínez, J., Barois, I. et al. (2012). Change in land use alters the diversity and composition of Bradyrhizobium communities and led to the introduction of Rhizobium etli into the tropical rain forest of Los Tuxtlas (Mexico). Microbial Ecology, 63, 822–834.
Ormeño-Orrillo, E., Rogel, M. A., Lloret, L., López, A., Martínez, J., Vinuesa, P. et al. (2009). Rhizobial diversity in different land use systems in the rain forest of Los Tuxtlas, Mexico. En I. Barois, E. J. Huising, P. Okoth, D. Trejo y M. De Los Santos (Eds.), Below-ground biodiversity in Sierra Santa Marta, Los Tuxtlas, Veracruz, México (pp. 65–84). Xalapa, Veracruz: Instituto de Ecología, A.C.
Ormeño-Orrillo, E., Servín-Garcidueñas, L. E., Rogel, M. A., González, V., Peralta, H., Mora, J. et al. (2015). Taxonomy of rhizobia and agrobacteria from the Rhizobiaceae family in light of genomics. Systematic and Applied Microbiology, 38, 287–291.
Ormeño-Orrillo, E., Vinuesa, P., Zúñiga-Dávila, D. y Martínez-Romero, E. (2006). Molecular diversity of native bradyrhizobia isolated from lima bean (Phaseolus lunatus L.) in Peru. Systematic and Applied Microbiology, 29, 253–262.
Parker, M. A. (2002). Bradyrhizobia from wild Phaseolus, Desmodium, and Macroptilium species in Northern Mexico. Applied and Environmental Microbiology, 68, 2044–2048.
Parker, M. A. (2004). rRNA and dnaK relationships of Bradyrhizobium sp. nodule bacteria from four Papilionoid legume trees in Costa Rica. Systematic and Applied Microbiology, 27, 334–342.
Peralta, H., Aguilar, A., Díaz, R., Mora, Y., Martínez-Batallar, G., Salazar, E. et al. (2016). Genomic studies of nitrogen-fixing rhizobial strains from Phaseolus vulgaris seeds and nodules. BMC Genomics, 17, 711.
Pérez-Ramírez, N. O., Rogel, M. A., Wang, E. T., Castellanos, J. Z. y Martínez-Romero, E. (1998). Seeds of Phaseolus vulgaris bean carry Rhizobium etli. FEMS Microbiology Ecology, 26, 289–296.
Piñero, D., Martínez, E. y Selander, R. K. (1988). Genetic diversity and relationships among isolates of Rhizobium leguminosarum biovar phaseoli. Applied and Environmental Microbiology, 54, 2825–2832.
Qian, J. H. y Parker, M. A. (2002). Contrasting nifD and ribosomal gene relationships among Mesorhizobium from Lotus oroboides in Northern Mexico. Systematic and Applied Microbiology, 25, 68–73.
Ramírez-Bahena, M. H., García-Fraile, P., Peix, A., Valverde, A., Rivas, R., Igual, J. M. et al. (2008). Revision of the taxonomic status of the species Rhizobium leguminosarum (Frank 1879) Frank 1889AL, Rhizobium phaseoli Dangeard 1926AL and Rhizobium trifolii Dangeard 1926AL. R. trifolii is a later synonym of R. leguminosarum. Reclassification of the strain R. leguminosarum DSM 30132 (= NCIMB 11478) as Rhizobium pisi sp. nov. International Journal of Systematic and Evolutionary Microbiology, 58, 2484–2490.
Ramírez-Puebla, S, T., Hernández-Rogel, M. A., Guerrero-Ruiz, G., Ormeño-Orrillo, E., Martínez-Romero, J. C., Servín-Garcidueñas, L. E. et al. (2018). Nodule bacteria from the cultured legume Phaseolus dumosus (belonging to the Phaseolus vulgaris cross-inoculation group) with common tropici phenotypic characteristics and symbiovar but distinctive phylogenomic position and chromid. Systematic and Applied Microbiology. doi: 10.1016/j.syapm.2018.12.007.
Reiter, B., Pfeifer, U., Schwab, H. y Sessitsch, A. (2002). Response of endophytic bacterial communities in potato plants to infection with Erwinia carotovora subsp. atroseptica. Applied and Environmental Microbiology, 68, 2261–2268.
Ribeiro, R. A., Martins, T. B., Ormeño-Orrillo, E., Marçon Delamuta, J. R., Rogel, M. A., Martínez-Romero, E. et al. (2015). Rhizobium ecuadorense sp. nov., an indigenous N2-fixing symbiont of the Ecuadorian common bean (Phaseolus vulgaris L.) genetic pool. International Journal of Systematic and Evolutionary Microbiology, 65, 3162–3169.
Ribeiro, R. A., Ormeño-Orrillo, E., Dall’Agnol, R. F., Graham, P. H., Martínez-Romero, E. y Hungria, M. (2013). Novel Rhizobium lineages isolated from root nodules of the common bean (Phaseolus vulgaris L.) in Andean and Mesoamerican areas. Research in Microbiology, 164, 740–7488.
Ribeiro, R. A., Rogel, M. A., López-López, A., Ormeño-Orrillo, E., Barcellos, F. G., Martínez, J. et al. (2012). Reclassification of Rhizobium tropici type A strains as Rhizobium leucaenae sp. nov. International Journal of Systematic and Evolutionary Microbiology, 62, 1179–1184.
Richter, M. y Rosselló-Móra, R. (2009). Shifting the genomic gold standard for the prokaryotic species definition. Proceedings of the National Academy of Sciences USA, 106, 19126–19131.
Rincón-Rosales, R., Lloret, L., Ponce, E. y Martínez-Romero, E. (2009). Rhizobia with different symbiotic efficiencies nodulate Acaciella angustissima in Mexico, including Sinorhizobium chiapanecum sp. nov. which has common symbiotic genes with Sinorhizobium mexicanum. FEMS Microbiology Ecology, 67, 103–117.
Rincón-Rosales, R., Villalobos-Escobedo, J. M., Rogel, M. A., López-López, A., Martínez, J., Ormeño- Orrillo, E. et al. (2013). Rhizobium calliandrae, Rhizobium mayense and Rhizobium jaguaris, novel rhizobial species nodulating the medicinal legume Calliandra grandiflora. International Journal of Systematic and Evolutionary Microbiology, 63, 3423–3429.
Robledo, M., Velázquez, E., Ramírez-Bahena, M. H., García-Fraile, P., Pérez-Alonso, A., Rivas, R. et al. (2011). The celC gene, a new phylogenetic marker useful for taxonomic studies in Rhizobium. Systematic and Applied Microbiology, 34, 393–399.
Rogel, M. A., Bustos, P., Santamaría, R. I., González, V., Romero, D., Cevallos, M. Á. et al. (2014). Genomic basis of symbiovar mimosae in Rhizobium etli. BMC Genomics, 15, 575.
Rogel, M. A., Hernández-Lucas, I., Kuykendall, L. D., Balkwill, D. L. y Martínez-Romero, E. (2001). Nitrogen-fixing nodules with Ensifer adhaerens harboring Rhizobium tropici symbiotic plasmids. Applied and Environmental Microbiology, 67, 3264–3268.
Rogel, M. A., Ormeño-Orrillo, E. y Martínez-Romero, E. (2011). Symbiovars in rhizobia reflect bacterial adaptation to legumes. Systematic and Applied Microbiology, 34, 96–104.
Rosenblueth, M. y Martínez-Romero, E. (2004). Rhizobium etli maize populations and their competitiveness for root colonization. Archives of Microbiology, 181, 337–344.
Rosenblueth, M. y Martínez-Romero, E. (2006). Bacterial endophytes and their interactions with hosts. Molecular Plant Microbe Interactions, 19, 827–837.
Ruiz-Valdiviezo, V. M., Rogel-Hernandez, M. A., Guerrero, G., Rincón-Molina, C. I., García-Pérez, L. G., Gutiérrez-Miceli, F. A. et al. (2017). Complete genome sequence of a novel nonnodulating Rhizobium species isolated from Agave americana L. rhizosphere. Genome Announcements, 5, e01280-17.
Santamaría, R. I., Bustos, P., Pérez-Carrascal, O. M., Miranda-Sánchez, F., Vinuesa, P., Martínez-Flores, I. et al. (2017). Complete genome sequences of eight Rhizobium symbionts associated with common bean (Phaseolus vulgaris). Genome Announcements, 5, e00645-17.
Savci, S. (2012). An agricultural pollutant: Chemical fertilizer. International Journal of Environmental Science and Development, 3, 73–80.
Segovia, L., Young, J. P. W. y Martínez-Romero, E. (1993). Reclassification of American Rhizobium leguminosarum biovar phaseoli type I strains as Rhizobium etli sp. nov. International Journal of Systematic Bacteriology, 43, 374–377.
Servín-Garcidueñas, L. E., Rogel, M. A., Ormeño-Orrillo, E., Zayas-Del Moral, A., Sánchez, F. y Martínez-Romero, E. (2016). Complete genome sequence of Bradyrhizobium sp. strain CCGE-LA001, isolated from field nodules of the enigmatic wild bean Phaseolus microcarpus. Genome Announcements, 4, e00126-16.
Servín-Garcidueñas, L. E., Zayas-Del Moral, A., Ormeño-Orrillo, E., Rogel, M. A., Delgado-Salinas, A., Sánchez, F. et al. (2014). Symbiont shift towards Rhizobium nodulation in a group of phylogenetically related Phaseolus species. Molecular Phylogenetics and Evolution, 79, 1–11.
Silva, C., Kan, F. L. y Martínez-Romero, E. (2007). Population genetic structure of Sinorhizobium meliloti and S. medicae isolated from nodules of Medicago spp. in Mexico. FEMS Microbiology Ecology, 60, 477–489.
Silva, C., Vinuesa, P., Eguiarte, L. E., Martínez-Romero, E. y Souza, V. (2003). Rhizobium etli and Rhizobium gallicum nodulate common bean (Phaseolus vulgaris) in a traditionally managed milpa plot in Mexico: population genetics and biogeographic implications. Applied and Environmental Microbiology, 69, 884–893.
Toledo, I., Lloret, L. y Martínez-Romero, E. (2003). Sinorhizobium americanum sp. nov., a new Sinorhizobium species modulating native Acacia spp. in Mexico. Systematic and Applied Microbiology, 26, 54–64.
Tong, W., Li, X., Huo, Y., Zhang, L., Cao, Y., Wang, E. et al. (2018). Genomic insight into the taxonomy of Rhizobium genospecies that nodulate Phaseolus vulgaris. Systematic and Applied Microbiology, 41, 300–310.
Vinuesa, P., Silva, C., Lorite, M. J., Izaguirre-Mayoral, M. L., Bedmar, E. J. y Martínez-Romero, E. (2005). Molecular systematics of rhizobia based on maximum likelihood and Bayesian phylogenies inferred from rrs, atpD, recA and nifH sequences, and their use in the classification of Sesbania microsymbionts from Venezuelan wetlands. Systematic and Applied Microbiology, 28, 702–716.
Wang, E. T., Kan, F. L., Tan, Z. Y., Toledo, I., Chen, W. X. y Martínez-Romero, E. (2003). Diverse Mesorhizobium plurifarium populations native to Mexican soils. Archives of Microbiology, 180, 444–454.
Wang, E. T., Rogel, M. A., García-de los Santos, A., Martínez-Romero, J., Cevallos, M. A. y Martínez-Romero, E. (1999). Rhizobium etli bv. mimosae, a novel biovar isolated from Mimosa affinis. International Journal of Systematic Bacteriology, 49, 1479–1491.
Wang, E. T., van Berkum, P., Beyene, D., Sui, X. H., Dorado, O., Chen, W. X. et al. (1998). Rhizobium huautlense sp. nov., a symbiont of Sesbania herbacea that has a close phylogenetic relationship with Rhizobium galegae. International Journal of Systematic Bacteriology, 48, 687–699.
Wang, F., Wang, E. T., Wu, L. J., Sui, X. H., Li Jr., Y. y Chen, W. X. (2011). Rhizobium vallis sp. nov., isolated from nodules of three leguminous species. International Journal of Systematic and Evolutionary Microbiology, 61, 2582–2588.
Young, J. P. W., Crossman, L. C., Johnston, A. W. B., Thomson, N. R., Ghazoui, Z. F., Hull, K. H. et al. (2006). The genome of Rhizobium leguminosarum has recognizable core and accessory components. Genome Biology, 7, R34.